公募研究(平成30年度~平成31年度)
| A01: 代謝アダプテーション |
イオウ代謝制御による骨格筋パフォーマンス改善とその分子機構の解明 | 研究代表者 | 本橋 ほづみ(東北大学) |
|---|---|---|---|
| 連携研究者 | 赤池 孝章(東北大学) | ||
| 細胞老化へ向かう代謝アダプテーションのエピゲノムからの探索 | 研究代表者 | 中山 啓子(東北大学) | |
| 脂肪細胞を用いた転写から翻訳までの統合的制御機構の理解 | 研究代表者 | 稲垣 毅(群馬大学) | |
| がん微小環境における代謝アダプテーションの解明 | 研究代表者 | 大澤 毅(東京大学) | |
| 連携研究者 | 神吉 康晴(東京大学) 松井 裕介(名古屋大学) 野中 綾(東京大学) |
||
| がん細胞の飛び道具「エクソソーム」のトランスオミクス解析 | 研究代表者 | 平山 明由(慶應義塾大学) | |
| 連携研究者 | 田畑 祥(慶應義塾大学) 森 大(慶應義塾大学) |
||
| 腸内微生物生態系が有する代謝アダプテーション機構の解明 | 研究代表者 | 福田 真嗣(慶應義塾大学) | |
| 連携研究者 | 馬場 健史(九州大学) 鈴木 健大(国立環境研究所) |
||
| 低体温と低酸素抵抗性を伴う生体保護代謝アダプテーションの解明 | 研究代表者 | 小早川 高(関西医科大学) | |
| 実験室進化による代謝アダプテーションの制約の解析 | 研究代表者 | 堀之内 貴明(理研) | |
| 連携研究者 | 古澤 力(理研) 前田 智也(理研) |
||
| 植物乾燥ストレス応答のトランスオミクス解析 | 研究代表者 | 平井 優美(理研) | |
| がん悪液質に対する代謝アダプテーションのトランスオミクス解析 | 研究代表者 | 河岡 慎平(京都大学) | |
| A02: トランスオミクス解析技術開発 |
生命現象の階層横断的な理解を可能する統計的モデリング技術の開発 | 研究代表者 | 島村 徹平(名古屋大学) |
| 連携研究者 | 鈴木 穣(東京大学) | ||
| キノームの機能制御メカニズムの解明 | 研究代表者 | 杉山 直幸(京都大学) | |
| データ駆動アプローチからのトランスオミクスネットワーク推定法の開発 | 研究代表者 | 宇田 新介(九州大学) | |
| 空間トランスオミクス技術の開発 | 研究代表者 | 大川 恭行(九州大学) |
※第二期(令和2年度~令和3年度)の公募研究班はこちらです。
A01: 代謝アダプテーション
イオウ代謝による骨格筋パフォーマンス改善とその分子機構の解明
 研究代表者:本橋 ほづみ(東北大学・加齢医学研究所・教授)
研究代表者:本橋 ほづみ(東北大学・加齢医学研究所・教授)
連携研究者:赤池 孝章(東北大学・大学院医学系研究科・教授)
HPhttp://www2.idac.tohoku.ac.jp/dep/ger/
概要
 骨格筋パフォーマンス改善におけるKEAP1-NRF2ストレス応答系とCARS2-SQR-ETHE1イオウ代謝制御系の機能的相互作用を明らかにするとともに、習慣的運動に誘導される代謝変化が遺伝子発現変化やタンパク質の機能変化を介して骨格筋を新たな定常状態へ遷移させる分子機構を明らかにする。前者では、34Sシステインを用いてミトコンドリアにおけるイオウ代謝のフラックス解析を実施し、イオウ代謝のキネティクスを明らかにする。そこから、NRF2の活性化によるイオウ代謝促進の作用点を推測するとともに、NRF2標的遺伝子の中から責任因子をスクリーニングにより同定する。後者では、習慣的運動をさせながら飼育した野生型マウスと全身でNRF2が活性化しているKeap1ノックダウンマウスを、それぞれのsedentary controlと比較して、血漿・骨格筋におけるイオウ代謝フラックスの変化、中心代謝物の変化、遺伝子発現の変化、エピゲノムの変化、タンパク質翻訳の変化を調べ、トランスオミクス解析により習慣的運動がもたらすイオウ代謝適応の全体像を明らかにする。
骨格筋パフォーマンス改善におけるKEAP1-NRF2ストレス応答系とCARS2-SQR-ETHE1イオウ代謝制御系の機能的相互作用を明らかにするとともに、習慣的運動に誘導される代謝変化が遺伝子発現変化やタンパク質の機能変化を介して骨格筋を新たな定常状態へ遷移させる分子機構を明らかにする。前者では、34Sシステインを用いてミトコンドリアにおけるイオウ代謝のフラックス解析を実施し、イオウ代謝のキネティクスを明らかにする。そこから、NRF2の活性化によるイオウ代謝促進の作用点を推測するとともに、NRF2標的遺伝子の中から責任因子をスクリーニングにより同定する。後者では、習慣的運動をさせながら飼育した野生型マウスと全身でNRF2が活性化しているKeap1ノックダウンマウスを、それぞれのsedentary controlと比較して、血漿・骨格筋におけるイオウ代謝フラックスの変化、中心代謝物の変化、遺伝子発現の変化、エピゲノムの変化、タンパク質翻訳の変化を調べ、トランスオミクス解析により習慣的運動がもたらすイオウ代謝適応の全体像を明らかにする。
参考文献
- Akaike, T., Ida, T., Wei, F.-Y., Nishida, M., Kumagai, Y., Alam, M.M., Ihara, H., Sawa, T., Matsunaga, T., Kasamatsu, S., Nishimura, A., Morita, M., Tomizawa, K., Nishimura, A., Watanabe, S., Inaba, K., Shima, H., … Motohashi, H. Cysteinyl-tRNA synthetase governs cysteine polysulfidation and mitochondrial bioenergetics. Nat. Commun. 8, 1177 (2017).
- Kitamura, H., Onodera, Y., Murakami, S., Suzuki, T., and Motohashi, H. IL-11 contribution to tumorigenesis in an NRF2 addiction cancer model. Oncogene. 36, 6315-6324 (2017).
- Mitsuishi, Y., Taguchi, K., Kawatani, Y., Shibata, T., Nukiwa, T., Aburatani, H., Yamamoto, M., and Motohashi, H. Nrf2 redirects glucose and glutamine into anabolic pathways in metabolic reprogramming. Cancer Cell. 22, 66-79 (2012).
A01: 代謝アダプテーション
細胞老化へ向かう代謝アダプテーションのエピゲノムからの探索
 研究代表者:中山 啓子(東北大学・大学院医学系研究科・教授)
研究代表者:中山 啓子(東北大学・大学院医学系研究科・教授)
HPhttp://www.devgen.med.tohoku.ac.jp/
概要
RASシグナルに応答して細胞は、老化の表現型を示し増殖を停止する。RASシグナルというシンプルな経路が代謝を老化状態へと変化させるメカニズムを、トランスクリプトームとエピゲノムのトランスオミクスの観点から解明することが本研究の目的である。
RASは細胞の増殖を制御するシグナル伝達分子である。RASシグナル伝達経路(以下、RASシグナル)の異常な活性化は細胞老化を引き起こすが、これは細胞増殖の不可逆的な停止やミトコンドリア機能の亢進といった、代謝状態の大きな変化を伴う過程である。RASシグナルの活性化は5日以内に細胞増殖やミトコンドリア機能に関わる遺伝子の発現を変化させる。しかしながら、細胞のRAS遺伝子に突然変異が起きた時に想定される長期間にわたる持続的なRASシグナルの活性化が、細胞の代謝を老化状態へと適応させるメカニズムは不明である。これまでに申請者は、1) RASシグナルの活性変化は短期間(3日間)にさまざまな遺伝子の転写状態を変化させるが、この時点ではヒストンのエピゲノムは大きく変化しないこと、2) 持続的なRASシグナルにより転写状態の変化が継続すると、それに伴ってエピゲノムも長期間(35日間)にわたりゆっくりと変化し続けることを見出している。このような段階的な変化は、RASシグナルに応答して老化する細胞の過渡状態と適応状態を反映していると考えられる。
長期間にわたる持続的なRASシグナルがエピゲノムを変化させる分子機構を解明し、持続的なRASシグナルに応答したエピゲノムの変化が、細胞の代謝を老化状態へと適応させるうえで果たす役割を明らかにする。

参考文献
- Hosogane, M., Funayama, R., Shirota, M., and Nakayama, K. Lack of Transcription Triggers H3K27me3 Accumulation in the Gene Body. Cell Rep. 16, 696-706 (2016).
- Nakagawa, T., Hosogane, M., Nakagawa, M., Morohoshi, A., Funayama, R., and Nakayama, K. Transforming Growth Factor β-Induced Proliferative Arrest Mediated by TRIM26-Dependent TAF7 Degradation and Its Antagonism by MYC. Mol. Cell. Biol. 38, e00449-17 (2018).
- Hosogane, M., Funayama, R., Nishida, Y., Nagashima, T., and Nakayama, K. Ras-Induced Changes in H3K27me3 Occur after Those in Transcriptional Activity. Barsh GS, ed. PLoS Genet. 9, e1003698 (2013).
A01: 代謝アダプテーション
脂肪細胞を用いた転写から翻訳までの統合的制御機構の理解
 研究代表者:稲垣 毅(群馬大学・生体調節研究所・教授
研究代表者:稲垣 毅(群馬大学・生体調節研究所・教授
HPhttp://epigenetics.imcr.gunma-u.ac.jp
概要
 細胞は外部刺激を感知して分化や形質転換をはじめとするさまざまな環境応答を行う。このような細胞活動の多くは転写や翻訳を介するものであり、エネルギー消費をともなう過程である。そのため、細胞内のエネルギー状態を感知して転写や翻訳を制御する機構が必要であると考えられる。エネルギー欠乏状態の細胞では転写や翻訳を極力抑える一方、エネルギー供給が保たれた細胞では転写や翻訳を活発化する余地がある。たとえば寒冷環境では、エネルギー蓄積型の白色脂肪がベージュ脂肪に形質転換し、熱産生を活発化させる。本研究ではこのような脂肪細胞の可塑性に注目し、脂肪細胞をモデルとしてエネルギー代謝物濃度解析とエネルギー代謝状態を記憶するエピゲノムメチローム修飾解析、トランスクリプトーム解析、タンパク発現解析を実施し、統合的な解析を行う。その結果、脂肪細胞の性質制御に関わる新規機構の解明を目指す。
細胞は外部刺激を感知して分化や形質転換をはじめとするさまざまな環境応答を行う。このような細胞活動の多くは転写や翻訳を介するものであり、エネルギー消費をともなう過程である。そのため、細胞内のエネルギー状態を感知して転写や翻訳を制御する機構が必要であると考えられる。エネルギー欠乏状態の細胞では転写や翻訳を極力抑える一方、エネルギー供給が保たれた細胞では転写や翻訳を活発化する余地がある。たとえば寒冷環境では、エネルギー蓄積型の白色脂肪がベージュ脂肪に形質転換し、熱産生を活発化させる。本研究ではこのような脂肪細胞の可塑性に注目し、脂肪細胞をモデルとしてエネルギー代謝物濃度解析とエネルギー代謝状態を記憶するエピゲノムメチローム修飾解析、トランスクリプトーム解析、タンパク発現解析を実施し、統合的な解析を行う。その結果、脂肪細胞の性質制御に関わる新規機構の解明を目指す。
参考文献
- Inagaki, T., Sakai, J., and Kajimura, S. Transcriptional and epigenetic control of brown and beige adipose cell fate and function. Nat. Rev. Mol. Cell Biol. 17, 480-495 (2016).
- Abe, Y., Rozqie, R., Matsumura, Y., Kawamura, T., Nakaki, R., Tsurutani, Y., Tanimura-Inagaki, K., Shiono, A., Magoori, K., Nakamura, K., Ogi, S., Kajimura, S., Kimura, H., Tanaka, T., Fukami, K., Osborne, T.F., Kodama, T., … Inagaki, T.*, and Sakai, J.* JMJD1A is a signal-sensing scaffold that regulates acute chromatin dynamics via SWI/SNF association for thermogenesis. Nat. Commun. 6, 7052 (2015).
- Inagaki, T., Iwasaki, S., Matsumura, Y., Kawamura, T., Tanaka, T., Abe, Y., Yamasaki, A., Tsurutani, Y., Yoshida, A., Chikaoka, Y., Nakamura, K., Magoori, K., Nakaki, R., Osborne, T.F., Fukami, K., Aburatani, H., Kodama, T., and Sakai, J. The FBXL10/KDM2B scaffolding protein associates with novel polycomb repressive complex-1 to regulate adipogenesis. J. Biol. Chem. 290, 4163-4177 (2015).
A01: 代謝アダプテーション
がん微小環境における代謝アダプテーションの解明

| 研究代表者: | 大澤 毅(東京大学・先端科学技術研究センター・特任准教授) |
| 連携研究者: | 神吉 康晴 (東京大学・アイソトープ総合センター・助教) 松井 裕介(名古屋大学・医学系研究科・准教授) 野中 綾(東京大学・先端科学技術研究センター・特任研究員) |
概要
がんの悪性化には過酷な腫瘍微小環境に適応する多重のがん代謝システム、すなわち「代謝アダプテーション」が重要な役割を果たす。申請者は、これまでゲノム・エピゲノム・トランスクリプトーム・メタボロームのトランスオミクス解析から「腫瘍微小環境」という過酷な環境変化に適応して、がん悪性化や治療抵抗性を促進すること、① エピゲノム因子ががん悪性化を促進すること、② 低pHでSREBP2が酢酸代謝を制御しがんの進展に関与すること、③ 低栄養で誘導される長鎖非コードRNA(LncRNA)ががんの悪性化を促進することを見出し報告してきた。
本研究はこれまで培った腫瘍微小環境におけるトランスオミクス解析をさらに発展させ、「代謝アダプテーション」の観点からがん悪性化の解明と治療法の開発を目指す。腫瘍微小環境の「代謝適応フィードバックシステム」という新概念から新規癌制御法の開発と、現存する化学療法との併用で相乗効果が期待できる標的分子の探索など治療への応用のための基盤となる研究が期待できる。
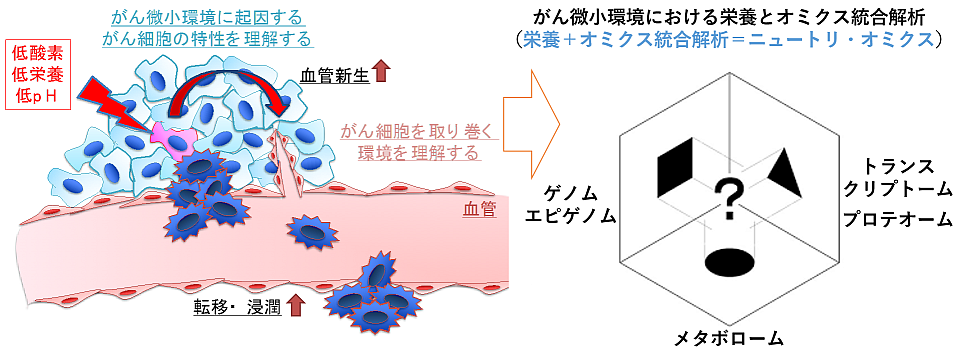
参考文献
- Kondo, A., Nonaka, A., Shimamura, T., Yamamoto, S., Yoshida, T., Kodama, T., Aburatani, H., and Osawa, T. Long Noncoding RNA JHDM1D-AS1 Promotes Tumor Growth by Regulating Angiogenesis in Response to Nutrient Starvation. Mol. Cell. Biol. 37, e00125-17 (2017).
- Kanki, Y., Nakaki, R., Shimamura, T., Matsunaga, T., Yamamizu, K., Katayama, S., Suehiro, J., Osawa, T., Aburatani, H., Kodama, T., Wada, Y., Yamashita, J.K., and Minami, T. Dynamically and epigenetically coordinated GATA/ETS/SOX transcription factor expression is indispensable for endothelial cell differentiation. Nucleic Acids Res. 45, 4344-4358 (2017).
- Kondo, A., Yamamoto, S., Nakaki, R., Shimamura, T., Hamakubo, T., Sakai, J., Kodama, T., Yoshida, T., Aburatani, H., and Osawa, T. Extracellular Acidic pH Activates the Sterol Regulatory Element-Binding Protein 2 to Promote Tumor Progression. Cell Rep. 18, 2228-2242 (2017).
A01: 代謝アダプテーション
がん細胞の飛び道具「エクソソーム」のトランスオミクス解析

| 研究代表者: | 平山 明由(慶應義塾大学・先端生命科学研究所・特任講師) |
| 連携研究者: | 田畑 祥(慶應義塾大学・先端生命科学研究所・特任講師) 森 大(慶應義塾大学・先端生命科学研究所・特任助教) |
HPhttp://metabolome.iab.keio.ac.jp/ja/
概要
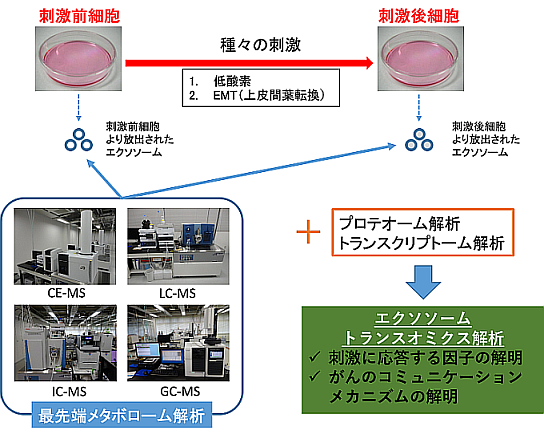 エクソソームは、体内の様々な細胞から分泌される直径30~100 nmの脂質二重膜構造を有する小胞である。エクソソームの発見当初は、細胞内の不要物を外に排出するごみ箱の役割を担っていると推測されていたが、エクソソーム内に存在するmiRNAの発見以降、新しい細胞間情報伝達システムとしてエクソソームが注目されている。本研究では、最先端のメタボローム解析技術を駆使し、これまで詳細に解析されてこなかった、がんエクソソーム中に含まれる代謝物の全容を明らかにする。また、がん悪性化に関与する低酸素刺激や上皮間葉転換(EMT)をモデルとして、エクソソームに含まれるRNA、タンパク質、代謝物等の構成要素が他の細胞にどのような影響を与えるのかをトランスオミクス解析によって解明することを目指す。これにより、エクソソーム中の代謝物の全容が解明されるとともに、エクソソームを介したがん細胞間のコミュニケーションの一端を解明できることが期待される。
エクソソームは、体内の様々な細胞から分泌される直径30~100 nmの脂質二重膜構造を有する小胞である。エクソソームの発見当初は、細胞内の不要物を外に排出するごみ箱の役割を担っていると推測されていたが、エクソソーム内に存在するmiRNAの発見以降、新しい細胞間情報伝達システムとしてエクソソームが注目されている。本研究では、最先端のメタボローム解析技術を駆使し、これまで詳細に解析されてこなかった、がんエクソソーム中に含まれる代謝物の全容を明らかにする。また、がん悪性化に関与する低酸素刺激や上皮間葉転換(EMT)をモデルとして、エクソソームに含まれるRNA、タンパク質、代謝物等の構成要素が他の細胞にどのような影響を与えるのかをトランスオミクス解析によって解明することを目指す。これにより、エクソソーム中の代謝物の全容が解明されるとともに、エクソソームを介したがん細胞間のコミュニケーションの一端を解明できることが期待される。
参考文献
- Hirayama, A., Abe, H., Yamaguchi, N., Tabata, S., Tomita, M., and Soga, T. Development of a sheathless CE-ESI-MS interface. Electrophoresis. March 2018
- Krycer, J.R., Yugi, K., Hirayama, A., Fazakerley, D.J., Quek, L.-E., Scalzo, R., Ohno, S., Hodson, M.P., Ikeda, S., Shoji, F., Suzuki, K., Domanova, W., Parker, B.L., Nelson, M.E., Humphrey, S.J., Turner, N., Hoehn, K.L., … James, D.E. Dynamic Metabolomics Reveals that Insulin Primes the Adipocyte for Glucose Metabolism. Cell Rep. 21, 3536-3547 (2017).
- Hirayama, A., Igarashi, K., Tomita, M., and Soga, T. Development of quantitative method for determination of γ-glutamyl peptides by capillary electrophoresis tandem mass spectrometry: An efficient approach avoiding matrix effect. J. Chromatogr. A. 1369, 161-169 (2014).
A01: 代謝アダプテーション
腸内微生物生態系が有する代謝アダプテーション機構の解明

| 研究代表者: | 福田 真嗣(慶應義塾大学・先端生命科学研究所・特任教授) |
| 連携研究者: | 馬場 健史(九州大学・生体防御医学研究所・教授) 鈴木 健大(国立環境研究所・生物・生態系環境研究センター・特別研究員) |
HPhttps://vu.sfc.keio.ac.jp/faculty_profile/cgi/f_profile.cgi?id=4b6eeaf56e240ec4
概要
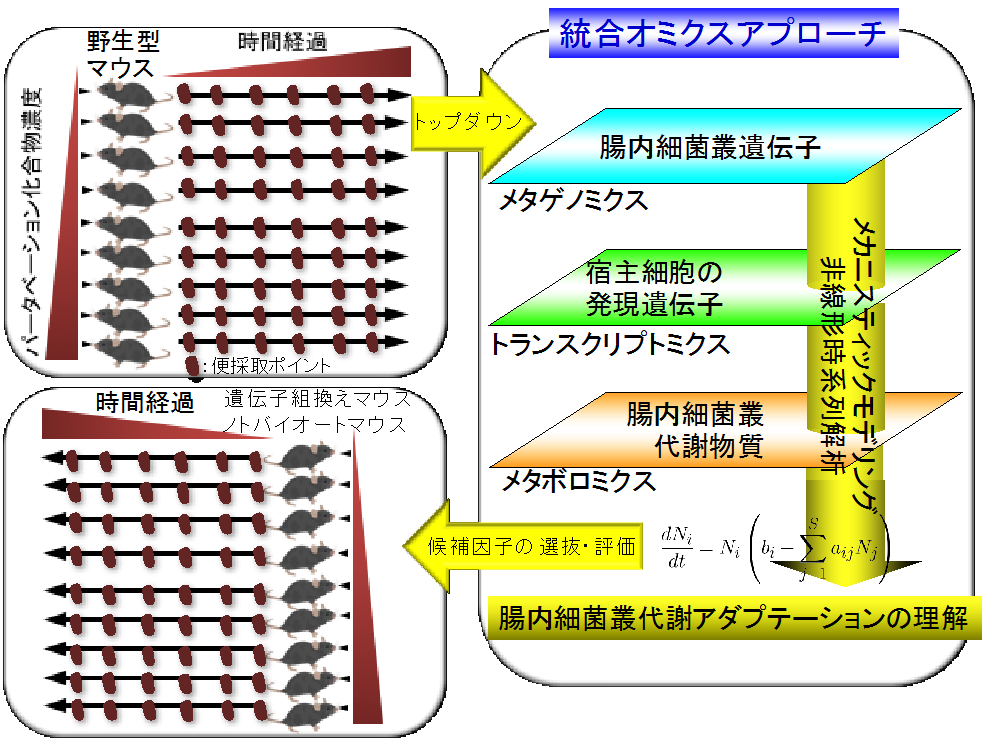 ヒトの腸管内には数百種類以上でおよそ100兆個もの腸内細菌が生息しており、これら腸内細菌叢は宿主腸管細胞と密接に相互作用することで、複雑な腸内生態系を形成している。われわれはこれまでに、メタボロミクスを基盤とする統合オミクス解析手法を構築し、腸内細菌叢から産生される代謝物質が、宿主の腸管局所のみならず、全身の恒常性維持に重要であることを明らかにした1-3。腸内細菌叢の機能が明らかになるにつれて、その次の課題として、複雑な腸内細菌叢全体の代謝機能をどのように制御するか、すなわち異種生物で構成される複雑な腸内微生物生態系の代謝アダプテーションの理解とその制御という課題があげられる。そこで本研究では、これまで異種生物と考えられてきた腸内細菌叢を一つの臓器として捉え、食事内容や薬剤投与などの環境因子パータベーションを与えた際の腸内環境変動について、統合オミクス解析や数理生物学的解析などの学際的研究アプローチにより、腸内細菌叢が有する代謝アダプテーション機構の理解を目指す。
ヒトの腸管内には数百種類以上でおよそ100兆個もの腸内細菌が生息しており、これら腸内細菌叢は宿主腸管細胞と密接に相互作用することで、複雑な腸内生態系を形成している。われわれはこれまでに、メタボロミクスを基盤とする統合オミクス解析手法を構築し、腸内細菌叢から産生される代謝物質が、宿主の腸管局所のみならず、全身の恒常性維持に重要であることを明らかにした1-3。腸内細菌叢の機能が明らかになるにつれて、その次の課題として、複雑な腸内細菌叢全体の代謝機能をどのように制御するか、すなわち異種生物で構成される複雑な腸内微生物生態系の代謝アダプテーションの理解とその制御という課題があげられる。そこで本研究では、これまで異種生物と考えられてきた腸内細菌叢を一つの臓器として捉え、食事内容や薬剤投与などの環境因子パータベーションを与えた際の腸内環境変動について、統合オミクス解析や数理生物学的解析などの学際的研究アプローチにより、腸内細菌叢が有する代謝アダプテーション機構の理解を目指す。
参考文献
- Kim, Y.-G., Sakamoto, K., Seo, S.-U., Pickard, J.M., Gillilland, M.G., Pudlo, N.A., Hoostal, M., Li, X., Wang, T.D., Feehley, T., Stefka, A.T., Schmidt, T.M., Martens, E.C., Fukuda, S., Inohara, N., Nagler, C.R., and Núñez, G. Neonatal acquisition of Clostridia species protects against colonization by bacterial pathogens. Science. 356, 315-319 (2017).
- Mishima, E.†, Fukuda, S.†(†equally contributed), Shima, H., Hirayama, A., Akiyama, Y., Takeuchi, Y., Fukuda, N.N., Suzuki, T., Suzuki, C., Yuri, A., Kikuchi, K., Tomioka, Y., Ito, S., Soga, T., and Abe, T. Alteration of the Intestinal Environment by Lubiprostone Is Associated with Amelioration of Adenine-Induced CKD. J. Am. Soc. Nephrol. 26, 1787-1794 (2015).
- Furusawa, Y.†, Obata, Y.†, Fukuda, S.†*(†equally contributed, *corresponding author), Endo, T.A., Nakato, G., Takahashi, D., Nakanishi, Y., Uetake, C., Kato, K., Kato, T., Takahashi, M., Fukuda, N.N., Murakami, S., Miyauchi, E., Hino, S., Atarashi, K., Onawa, S., … Hase, K.*, and Ohno, H.* Commensal microbe-derived butyrate induces the differentiation of colonic regulatory T cells. Nature. 504, 446-450 (2013).
A01: 代謝アダプテーション
低体温と低酸素抵抗性を伴う生体保護代謝アダプテーションの解明
 研究代表者:小早川 高(関西医科大学・生命医学研究所)
研究代表者:小早川 高(関西医科大学・生命医学研究所)
概要
 恐怖は危機に瀕した個体を守る多様な行動や生理応答を誘発する強力な情動である。恐怖刺激が代謝状態を変動させるのか、もしそうであればその背後にある制御メカニズム、また、情動状態と代謝状態を結びつける生物学的な意義は未解明である。私たちは、嗅覚刺激による先天的と後天的な恐怖情報は鼻腔内で分離した神経回路により脳へ伝達されることを解明した(文献1、2)。さらに、分離して伝達された先天的と後天的な恐怖情報は恐怖中枢である扁桃体中心核のセロトニン2A受容体発現細胞において拮抗的に統合され、先天的な恐怖行動が後天的な恐怖行動に優先されるという階層制御を受けることを解明した(文献3)。これらの一連の研究の過程で、極めて強力な先天的恐怖情動を誘発する人工匂い分子群「チアゾリン類恐怖臭」(Thiazoline-related fear odors: tFOs)を開発した。本研究ではtFOsが誘発する「恐怖情動性代謝アダプテーション」を司る分子メカニズムを解明し、情動と代謝を結ぶ未知の原理の解明を目指す。
恐怖は危機に瀕した個体を守る多様な行動や生理応答を誘発する強力な情動である。恐怖刺激が代謝状態を変動させるのか、もしそうであればその背後にある制御メカニズム、また、情動状態と代謝状態を結びつける生物学的な意義は未解明である。私たちは、嗅覚刺激による先天的と後天的な恐怖情報は鼻腔内で分離した神経回路により脳へ伝達されることを解明した(文献1、2)。さらに、分離して伝達された先天的と後天的な恐怖情報は恐怖中枢である扁桃体中心核のセロトニン2A受容体発現細胞において拮抗的に統合され、先天的な恐怖行動が後天的な恐怖行動に優先されるという階層制御を受けることを解明した(文献3)。これらの一連の研究の過程で、極めて強力な先天的恐怖情動を誘発する人工匂い分子群「チアゾリン類恐怖臭」(Thiazoline-related fear odors: tFOs)を開発した。本研究ではtFOsが誘発する「恐怖情動性代謝アダプテーション」を司る分子メカニズムを解明し、情動と代謝を結ぶ未知の原理の解明を目指す。
参考文献
- Kobayakawa, K., Kobayakawa, R., Matsumoto, H., Oka, Y., Imai, T., Ikawa, M., Okabe, M., Ikeda, T., Itohara, S., Kikusui, T., Mori, K., and Sakano, H. Innate versus learned odour processing in the mouse olfactory bulb. Nature 450, 503-508 (2007).
- Matsuo, T., Hattori, T., Asaba, A., Inoue, N., Kanomata, N., Kikusui, T., Kobayakawa, R., and Kobayakawa, K. Genetic dissection of pheromone processing reveals main olfactory system-mediated social behaviors in mice. Proc. Natl. Acad. Sci. U. S. A. 112, E311-320 (2015).
- Isosaka, T., Matsuo, T., Yamaguchi, T., Funabiki, K., Nakanishi, S., Kobayakawa, R., and Kobayakawa, K. Htr2a-Expressing Cells in the Central Amygdala Control the Hierarchy between Innate and Learned Fear. Cell 163, 1153-1164 (2015).
A01: 代謝アダプテーション
実験室進化による代謝アダプテーションの制約の解析

| 研究代表者: | 堀之内 貴明(理化学研究所・生命機能科学研究センター・研究員) |
| 連携研究者: | 古澤 力(理化学研究所・生命機能科学研究センター・チームリーダー) 前田 智也(理化学研究所・生命機能科学研究センター・基礎科学特別研究員) |
HPhttps://sites.google.com/site/takaakihorinouchi/
概要
本研究では、代謝アダプテーションの過程において取り得る表現型が一定の範囲に制約されるメカニズムを解明し、その予測と制御を可能とする手法を開発する。生物システムは環境変動に対し、その表現型を柔軟に変化させる(代謝アダプテーション)。しかし、その代謝アダプテーションによる表現型の変化は変幻自在というわけではなく、取り得る表現型には制約が存在する。さまざまな実験研究がそうした制約の存在を示しているが、そのメカニズムの多くは不明であり、取り得る表現型の範囲も明確にはなっていない。そこで本研究では、さまざまな代謝阻害剤を用いた大腸菌の実験室進化を用い、その代謝アダプテーションの過程における表現型変化を定量する。得られた高次元データを少数の自由度で適切に記述する数理モデルを構築することにより、代謝アダプテーションがどのような制約を受けているかを明らかにし、その背後にあるメカニズムの解明を行う。さらに、その解析に基づいて代謝アダプテーションの過程を予測および操作する手法の開発と実験的検証を行う。
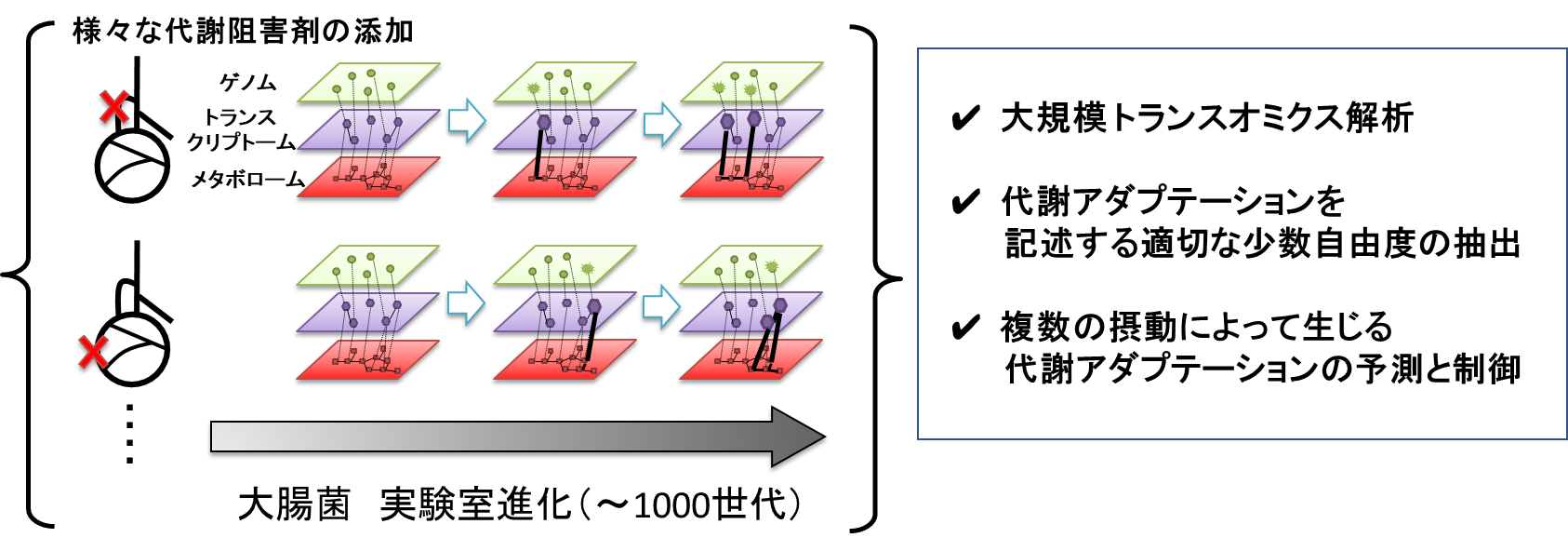
参考文献
- Tokuyama, K., Toya, Y., Horinouchi, T., Furusawa, C., Matsuda, F., and Shimizu, H. Application of adaptive laboratory evolution to overcome a flux limitation in an Escherichia coli production strain. Biotechnol. Bioeng. 115, 1542-1551 (2018).
- Horinouchi, T., Suzuki, S., Kotani, H., Tanabe, K., Sakata, N., Shimizu, H., and Furusawa, C. Prediction of Cross-resistance and Collateral Sensitivity by Gene Expression profiles and Genomic Mutations. Sci. Rep. 7, 14009 (2017).
- Horinouchi, T., Minamoto, T., Suzuki, S., Shimizu, H., and Furusawa, C. Development of an Automated Culture System for Laboratory Evolution. J. Lab. Autom. 19, 478-482 (2014).
A01: 代謝アダプテーション
植物乾燥ストレス応答のトランスオミクス解析
 研究代表者:平井 優美(理化学研究所・環境資源科学研究センター・チームリーダー)
研究代表者:平井 優美(理化学研究所・環境資源科学研究センター・チームリーダー)
HPhttp://www.csrs.riken.jp/jp/labs/msrt/index.html
概要
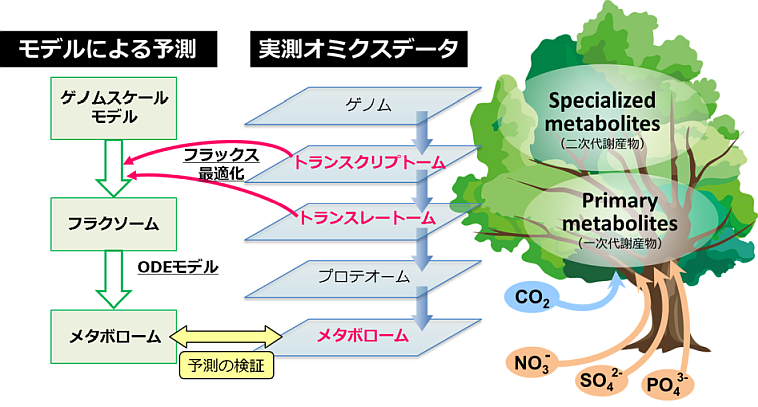
地面に根を下ろして動かない植物は静的なイメージがあるが、環境変化に応答、適応して生き延びる生存戦略を取っているためむしろ動物よりダイナミックな代謝アダプテーションの機構を有していると考えられる。炭素のフラックスだけに着目しても、光合成によりCO2を同化して作った糖を、成長段階や環境変動に応じてバイオマス、エネルギー、貯蔵、二次代謝へと自在に分配する精密な制御機構を植物が有していることがうかがえる。本研究では、乾燥ストレスに対する植物の代謝アダプテーションを対象にトランスオミクス解析を行う。モデル植物シロイヌナズナのゲノムスケールモデルを利用し、乾燥条件下のトランスクリプトームデータ、またはトランスレートームデータを用いてメタボロームの変化を予測する。実測によるメタボロームデータと予測結果を比較することで、mRNA量がフラックスの大きさを決定する代謝反応と翻訳量が決定する代謝反応を同定するとともに、植物の乾燥ストレス耐性の鍵となる代謝反応を明らかにする。
参考文献
- Sriyudthsak, K., Mejia, R.F., Arita, M., and Hirai, M.Y. PASMet: a web-based platform for prediction, modelling and analyses of metabolic systems. Nucleic Acids Res. 44, w205-W211 (2016).
- Sriyudthsak, K., Sawada, Y., Chiba, Y., Yamashita, Y., Kanaya, S., Onouchi, H., Fujiwara, T., Naito, S., Voit, E.O., Shiraishi, F., and Hirai, M.Y. A U-system approach for predicting metabolic behaviors and responses based on an alleged metabolic reaction network. BMC Syst. Biol. 8, s4 (2014).
- Sriyudthsak, K., Shiraishi, F., and Hirai, M.Y. Identification of a Metabolic Reaction Network from Time-Series Data of Metabolite Concentrations. Peddada SD, ed. PLoS One. 8, e51212 (2013).
A01: 代謝アダプテーション
がん悪液質に対する代謝アダプテーションのトランスオミクス解析
 研究代表者:河岡 慎平(京都大学・ウイルス・再生医科学研究所・特定准教授)
研究代表者:河岡 慎平(京都大学・ウイルス・再生医科学研究所・特定准教授)
HPhttps://sites.google.com/site/shinpeicharliekawaoka/
概要
がん悪液質は、がんが分泌するサイトカインやホルモンによって引き起こされる重度の全身性代謝障害の総称である。脂肪や筋肉、体重の減少がその代表的な症状として知られる。がん患者のQuality of Life (QOL) ならびに生命予後を著しく悪化させることから、がん悪液質を有効に制御する方法の開発が望まれている。しかしながら、がん悪液質のメカニズムは十分に理解されておらず、そのことが一因となり、現時点では、がん悪液質の有効な治療法は存在しない。研究代表者は、2014年以降、がん悪液質をはじめとするがんによる個体への悪影響にとって重要な宿主要因を同定することで、本問題の解決に貢献しようとしてきた (文献1&2)。本公募班では、これまでに得られた宿主因子に関する知見に代謝統合オミクスの視点を加えることで、がん悪液質を代謝アダプテーションの立場から捉えなおし、がん悪液質のメカニズムについての理解を前進させることを目指す。具体的には、担がんマウス個体の脂肪に対するトランスオミクス解析をおこない、脂肪で起こる遺伝子発現レベル・代謝物レベルでの変化を記載する。次に、数理モデル解析によって、遺伝子発現レベルの変化と代謝物レベルの変化をつなぐルールを発見する。さらに、これらの研究を通して、観測される「状態の変化」が、がんによる個体への悪影響の表れであるのか、それとも、がんに対する個体の対応 (アダプテーション) であるのか、を切り分け、がん悪液質というおそらくは非常に複雑な生理的異常のメカニズムをシンプルに整理するための一助とする。

参考文献
- Enya, S., Kawakami, K., Suzuki, Y., and Kawaoka, S. A novel zebrafish intestinal tumor model reveals a role for cyp7a1-dependent tumor-liver crosstalk in causing adverse effects on the host. Dis. Model. Mech. 11, dmm032383 (2018).
- Hojo, H., Enya, S., Arai, M., Suzuki, Y., Nojiri, T., Kangawa, K., Koyama, S., Kawaoka, S. Remote reprogramming of hepatic circadian transcriptome by breast cancer. Oncotarget. 8, 34128-34140 (2017).
A02: トランスオミクス解析技術開発
生命現象の階層横断的な理解を可能にする統計的モデリング技術の開発
 研究代表者:島村 徹平(名古屋大学・大学院医学系研究科・教授)
研究代表者:島村 徹平(名古屋大学・大学院医学系研究科・教授)
連携研究者:鈴木 穣(東京大学・大学院新領域創成科学研究科・教授)
HPhttp://www.nagoya-sysbiol.info/
概要
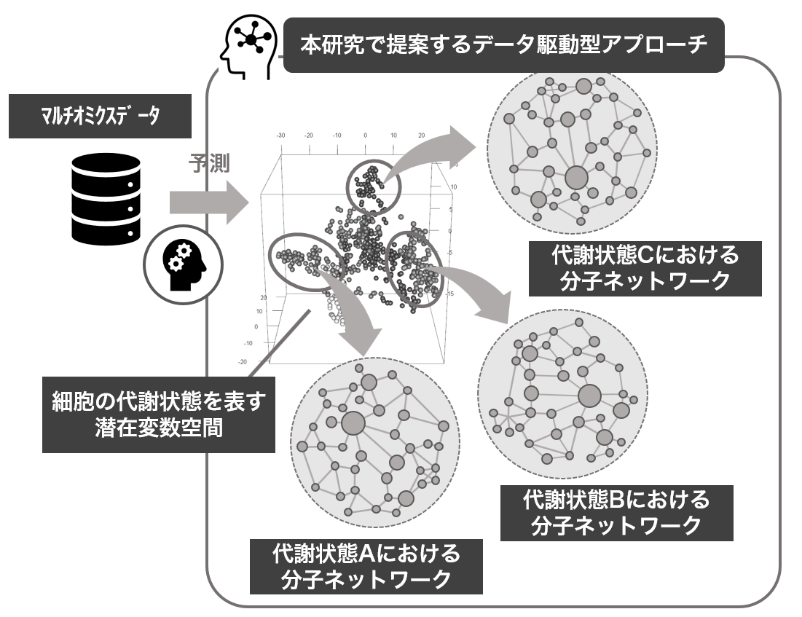 本研究課題では、さまざまなオミクスの階層横断的な情報統合を行い、代謝アダプテーションの分子機序を解明するための統計的モデリング技術を開発する。
本研究課題では、さまざまなオミクスの階層横断的な情報統合を行い、代謝アダプテーションの分子機序を解明するための統計的モデリング技術を開発する。
(1)細胞の代謝状態を予測する数理的アプローチの開発
メタボロームと他のオミクス情報が同時に観測されたデータから、細胞の代謝状態を表す代謝潜在変数を予測する数理アプローチを開発する。
(2)代謝状態の違いによるリモデリング機序を俯瞰するシステム生物学的アプローチの開発
細胞内外の代謝環境やエネルギー状態のダイナミックな変化が、細胞内外のシグナル伝達、エピゲノム制御、転写制御機構にどのような変化を引き起こすかを俯瞰するため、外部環境によってリモデリングされる分子ネットワークをオミクスデータから再構築するシステム生物学的手法を開発する。
計画班および公募班との有機的な連携のもと、代謝アダプテーションへの知見と理解を深め、階層横断的な情報統合を行う数理アプローチによる知識発見・仮説生成から生命科学のイノベーションを創出する情報解析基盤の構築を目指す。
参考文献
- Matsui, Y., Niida, A., Uchi, R., Mimori, K., Miyano, S., and Shimamura, T. phyC: Clustering cancer evolutionary trees. Markowetz F, ed. PLOS Comput. Biol. 13, e1005509 (2017).
- Uchi, R., Takahashi, Y., Niida, A., Shimamura, T., Hirata, H., Sugimachi, K., Sawada, G., Iwaya, T., Kurashige, J., Shinden, Y., Iguchi, T., Eguchi, H., Chiba, K., Shiraishi, Y., Nagae, G., Yoshida, K., Nagata, Y., … Mimori, K. Integrated Multiregional Analysis Proposing a New Model of Colorectal Cancer Evolution. Gerlinger M, ed. PLOS Genet. 12, e1005778 (2016).
- Matsui, Y., Mizuta, M., Ito, S., Miyano, S., and Shimamura, T. D 3 M: detection of differential distributions of methylation levels. Bioinformatics. 32, 2248-2255 (2016).
A02: トランスオミクス解析技術開発
キノームの機能制御メカニズムの解明
 研究代表者:杉山 直幸(京都大学・大学院薬学研究科・准教授)
研究代表者:杉山 直幸(京都大学・大学院薬学研究科・准教授)
HPhttp://www.pharm.kyoto-u.ac.jp/seizai/
概要
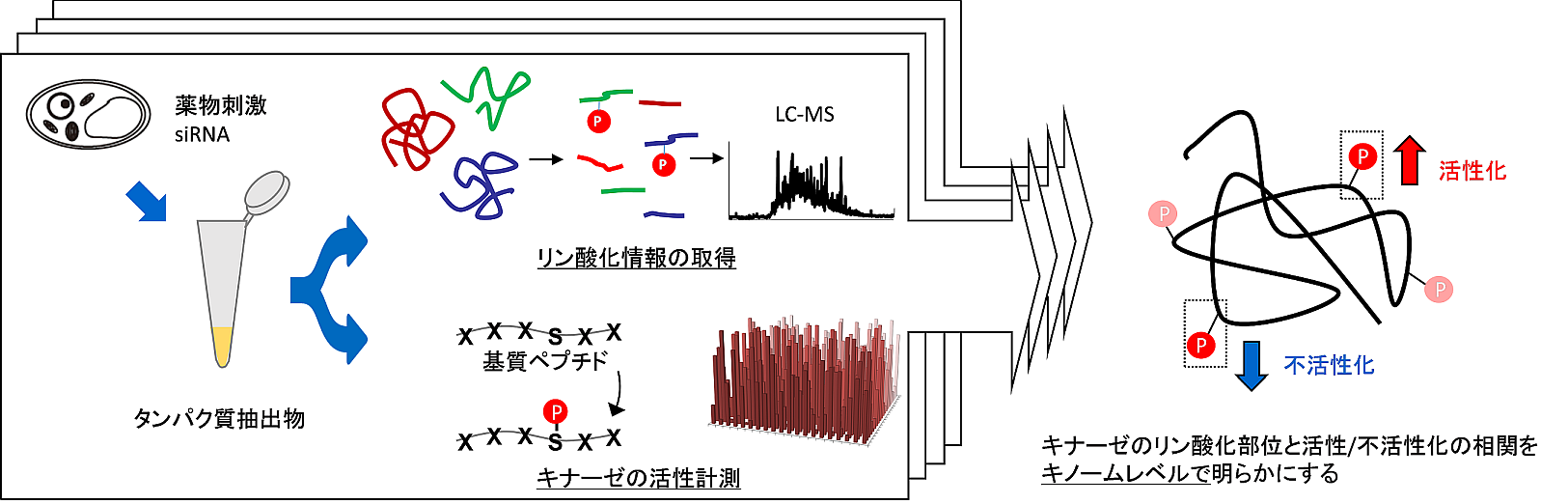
プロテインキナーゼに焦点を当て、そのキナーゼ活性とキナーゼ自身の翻訳後修飾との関連性、すなわちキナーゼの活性制御メカニズムをキノームレベルで一斉解析する手法を開発する。プロテインキナーゼによるタンパク質のリン酸化は真核生物の細胞内シグナル伝達において非常に重要な翻訳後修飾の一つであり、ヒトの場合少なくとも518種類の遺伝子がプロテインキナーゼをコードしていると考えられている。これらキナーゼの基質特異性や制御機構について多くの研究が行われているものの、解明されているものは一部であり、多くのキナーゼについては未解明のままである。本研究では、摂動を与えた細胞に対して、リン酸化プロテオミクスによるキナーゼ自身のリン酸化状態の取得とキナーゼ活性計測を同時に行い、キナーゼの翻訳後修飾と活性制御の関係を明らかにすることを目的とする。さらに、そのキナーゼ活性のスイッチON/OFFに関わる責任キナーゼをin vitroキナーゼ基質情報を基に予測し、領域内の翻訳後修飾プロテオミクスなどのデータと組み合わせることにより、包括的なシグナル伝達ネットワークを構築する。
参考文献
- Imamura, H., Wagih, O., Niinae, T., Sugiyama, N., Beltrao, P., and Ishihama, Y. Identifications of Putative PKA Substrates with Quantitative Phosphoproteomics and Primary-Sequence-Based Scoring. J. Proteome Res. 16, 1825-1830 (2017).
- Imamura, H., Sugiyama, N., Wakabayashi, M., and Ishihama, Y. Large-Scale Identification of Phosphorylation Sites for Profiling Protein Kinase Selectivity. J. Proteome Res. 13, 3410-3419 (2014).
- Sugiyama, N., Masuda, T., Shinoda, K., Nakamura, A., Tomita, M., and Ishihama, Y. Phosphopeptide enrichment by aliphatic hydroxy acid-modified metal oxide chromatography for nano-LC-MS/MS in proteomics applications. Mol. Cell. Proteomics. 6, 1103-1109 (2007).
A02: トランスオミクス解析技術開発
データ駆動アプローチからのトランスオミクスネットワーク推定法の開発
 研究代表者:宇田 新介(九州大学・生体防御医学研究所・准教授)
研究代表者:宇田 新介(九州大学・生体防御医学研究所・准教授)
HPhttp://www.bioreg.kyushu-u.ac.jp/labo/omics/index.html
概要
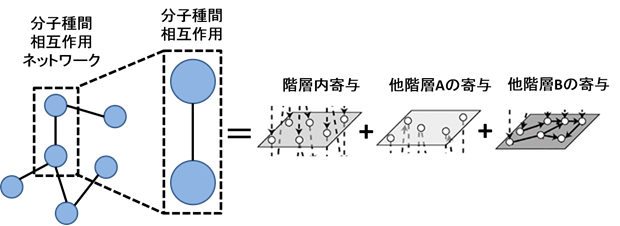
データ駆動アプローチから分子種間相互作用ネットワークの構造を推定する手法を開発し,実データへの適用を行う. 統計的条件付き独立性は,対象とする2変数以外の他の変数の影響を除いた2変数間の依存関係を示し,統計的な観点から変数間の相互作用を知るのに理想的な指標であるが,データが高次元である場合や標本サイズが小さい場合などには,実際の適用に際して困難が生じることが多く,特にオミクスデータではそれらの傾向が顕著になる.本研究計画では,情報理論と統計的検定の枠組みを用いて,高次元や標本サイズが比較的小さいトランスオミクスデータに対しても適用可能な統計的条件付き独立性を評価する手法を構築する.また,情報理論的解析から2分子種間の相互作用全体を遺伝子発現や代謝などの階層ごとの寄与に分解して解釈する.トランスオミクスデータの提供を受けて,開発手法をインスリン作用ネットワークの構造推定に適用する.
参考文献
- Uda, S., Saito, T.H., Kudo, T., Kokaji, T., Tsuchiya, T., Kubota, H., Komori, Y., Ozaki, Y., and Kuroda, S. Robustness and compensation of information transmission of signaling pathways. Science. 341, 558-561 (2013).
A02: トランスオミクス解析技術開発
空間トランスオミクス技術の開発
 研究代表者:大川 恭行(九州大学・生体防御医学研究所・教授)
研究代表者:大川 恭行(九州大学・生体防御医学研究所・教授)
HPhttp://tx.bioreg.kyushu-u.ac.jp/
概要
オミクス技術の発展は、ゲノム、エピゲノムからメタボロームに至るあらゆる細胞内の情報を1細胞レベルで高精度に解析することを目標に、改良、発展の道を辿ってきた。一方で、特定の組織形成や機能には、個々の細胞だけではなく、“隣り合う細胞”を含めた細胞同士の相互作用が欠かせず、従来Localization + ome:ローカライゾームとして提唱されてきた。しかしながら空間情報に紐づけされたオミクス解析技術の開発は殆ど行われていない。そこで、本研究では、組織内の細胞の位置情報を保持した状態での1細胞解像度の空間オミクス技術の開発を行う。まずトランスクリプトームとローカライゾームの同時解析法の確立を行い、続いてプロテオーム等の他のオームの同時解析技術を樹立させる。さらに次世代シークエンサー(NGS)の1回の解析で、同一細胞群のトランスオミクスを空間・位置情報に紐づけしつつ1細胞レベルで理解する新たなオミクス解析の発展を進める。現在1細胞エピゲノム技術ChILTを開発し、原理部分の動作可能性を実証している。本研究提案はこれらの技術面での優位点を活かし1細胞トランスオミクスと空間オミクスを同時に解析する技術の実現を目指す。
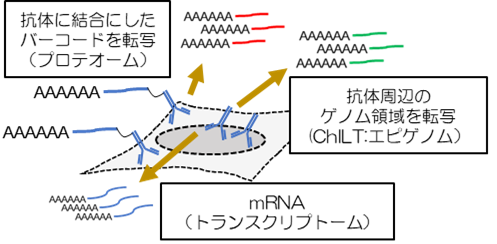
参考文献
- Harada, A., Maehara, K., Ono, Y., Taguchi, H., Yoshioka, K., Kitajima, Y., Xie, Y., Sato, Y., Iwasaki, T., Nogami, J., Okada, S., Komatsu, T., Semba, Y., Takemoto, T., Kimura, H., Kurumizaka, H., and Ohkawa, Y. Histone H3.3 sub-variant H3mm7 is required for normal skeletal muscle regeneration. Nat. Commun. 9, 1400 (2018).
- Semba, Y., Harada, A., Maehara, K., Oki, S., Meno, C., Ueda, J., Yamagata, K., Suzuki, A., Onimaru, M., Nogami, J., Okada, S., Akashi, K., and Ohkawa, Y. Chd2 regulates chromatin for proper gene expression toward differentiation in mouse embryonic stem cells. Nucleic Acids Res. 45, 8758-8772 (2017).
- Kudou, K., Komatsu, T., Nogami, J., Maehara, K., Harada, A., Saeki, H., Oki, E., Maehara, Y., and Ohkawa, Y. The requirement of Mettl3-promoted MyoD mRNA maintenance in proliferative myoblasts for skeletal muscle differentiation. Open Biol. 7, 170119 (2017).

