公募研究(令和2年度~令和3年度)
| A01: 代謝アダプテーション |
骨格筋の機能的・器質的リモデリングにおける硫黄代謝の貢献 | 研究代表者 | 本橋 ほづみ(東北大学) |
|---|---|---|---|
| がん微小環境における代謝適応フィードバックシステムの解明 | 研究代表者 | 大澤 毅(東京大学) | |
| 代謝入力による脂肪細胞の遺伝子発現機構の解明 | 研究代表者 | 酒井 寿郎(東京大学) | |
| メタボロミクス可視化センサーによるニューロンの代謝調節機構の解明 | 研究代表者 | 坪井 貴司(東京大学) | |
| 転写後遺伝子発現制御に着目した低酸素応答の代謝アダプテーションの機序解明 | 研究代表者 | 秋光 信佳(東京大学) | |
| 研究協力者 | 川村 猛(東京大学) 川田 健太郎(東京大学) |
||
| がんに起因する多臓器代謝異常に対する宿主アダプテーションのトランスオミクス解析 | 研究代表者 | 河岡 慎平(京都大学) | |
| 冬眠・人工冬眠・生命保護を結ぶ代謝アダプテーション | 研究代表者 | 小早川 令子(関西医科大学) | |
| 乾燥ストレスに対する植物の代謝アダプテーション機構の解明 | 研究代表者 | 平井 優美(理研) | |
| 線虫の幼虫期発生プログラムを起動する栄養シグナルと遺伝子発現ネットワークの解明 | 研究代表者 | 黒柳 秀人(琉球大学) | |
| A02: トランスオミクス解析技術開発 |
臓器連関トランスオミクスを読み解くベイズモデリング技術の開発 | 研究代表者 | 島村 徹平(名古屋大学) |
| 研究協力者 | 河岡 慎平(京都大学) | ||
| 摂動による迅速な翻訳応答を捉えるプロテオームリボソームプロファイリング法の開発 | 研究代表者 | 今見 考志(京都大学) | |
| 研究協力者 | 小形 公亮(京都大学) 石濱 泰(京都大学) |
||
| 活性制御部位関連ペプチドによるキノーム活性測定法の開発 | 研究代表者 | 杉山 直幸(京都大学) | |
| 高深度解析を可能とする単一細胞空間オミクス技術の開発 | 研究代表者 | 大川 恭行(九州大学) | |
| 情報量に基づいたトランスオミクスデータ解析手法の開発 | 研究代表者 | 宇田 新介(九州大学) | |
| テンソル分解を用いた教師無し学習による変数選択法を用いたトランスオミクス解析 | 研究代表者 | 田口 善弘(中央大学) | |
| 複数シグナル経路活性の同時計測を可能とする分子プローブ技術の開発 | 研究代表者 | 足達 俊吾(産総研) |
※第一期(平成30年度~平成31年度)の公募研究班はこちらです。
A01: 代謝アダプテーション
イオウ代謝による骨格筋パフォーマンス改善とその分子機構の解明
 研究代表者:本橋 ほづみ(東北大学・加齢医学研究所・教授)
研究代表者:本橋 ほづみ(東北大学・加齢医学研究所・教授)
HPhttp://www2.idac.tohoku.ac.jp/dep/ger/
概要
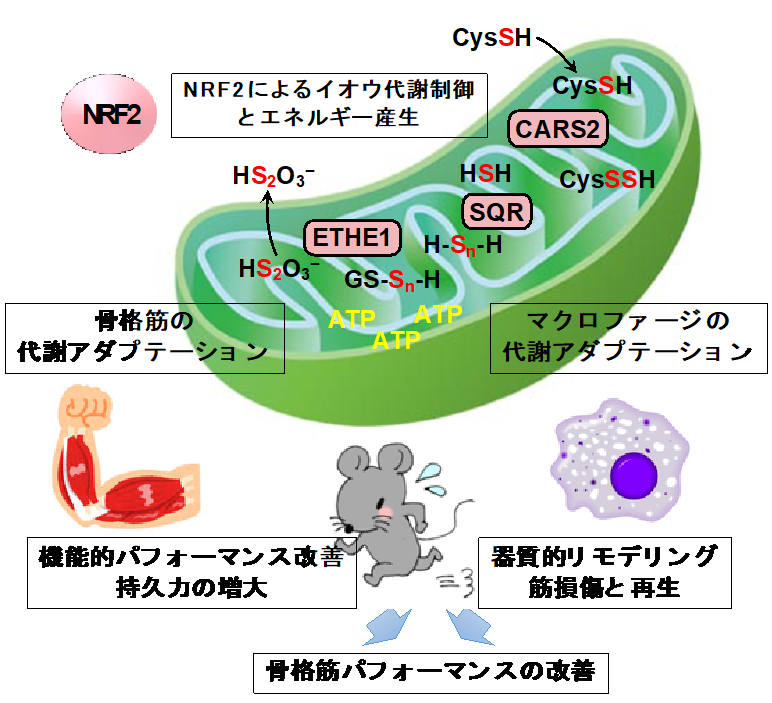 NRF2は生体防御において中心的役割を担う転写活性化因子であるが、最近、硫黄代謝を促進することにより、ミトコンドリア膜電位の維持に貢献することが明らかになった。そこで、本研究では、ミトコンドリアの硫黄依存性エネルギー代謝の生理的な意義と、NRF2の活性化がもたらすエネルギー代謝改善における活性硫黄種の貢献を明らかにすることを目的とする。特に、骨格筋の機能的なパーフォーマンス改善における代謝アダプテーションと、トレーニングによる骨格筋の器質的リモデリングを支えるマクロファージの代謝アダプテーションに着目して解析をすすめる。前者では骨格筋自体の代謝変化を中心に関連するオミックスデータを取得する。後者では、運動に伴う筋繊維損傷により動員されるマクロファージの機能がトレーニング効果を規定するとの認識に立ち、マクロファージの炎症応答におけるミトコンドリア代謝の改変と、それがもたらす遺伝子発現・エピゲノムの変化を解析する。そして、マクロファージでのNRF2活性化がもたらす硫黄代謝の促進が、筋繊維修復・再生に及ぼす影響を明らかにする。
NRF2は生体防御において中心的役割を担う転写活性化因子であるが、最近、硫黄代謝を促進することにより、ミトコンドリア膜電位の維持に貢献することが明らかになった。そこで、本研究では、ミトコンドリアの硫黄依存性エネルギー代謝の生理的な意義と、NRF2の活性化がもたらすエネルギー代謝改善における活性硫黄種の貢献を明らかにすることを目的とする。特に、骨格筋の機能的なパーフォーマンス改善における代謝アダプテーションと、トレーニングによる骨格筋の器質的リモデリングを支えるマクロファージの代謝アダプテーションに着目して解析をすすめる。前者では骨格筋自体の代謝変化を中心に関連するオミックスデータを取得する。後者では、運動に伴う筋繊維損傷により動員されるマクロファージの機能がトレーニング効果を規定するとの認識に立ち、マクロファージの炎症応答におけるミトコンドリア代謝の改変と、それがもたらす遺伝子発現・エピゲノムの変化を解析する。そして、マクロファージでのNRF2活性化がもたらす硫黄代謝の促進が、筋繊維修復・再生に及ぼす影響を明らかにする。
参考文献
- Saigusa, D., Motoike, U., Saito, S., Zorzi, M., Aoki, Y., Kitamura, H., Suzuki, M., Katsuoka, F., Ishii, H., Kinoshita, K., Motohashi, H. & Yammaoto M. Impacts of NRF2 Activation in Non-Small-Cell Lung Cancer Cell Lines on Extracellular Metabolites. Cancer Sci. 111, 667-678 (2020).
- Motohashi, H. & Akaike, T. Sulfur-utilizing cytoprotection and energy metabolism. Curr. Opin. Physhiol. 9, 1-8 (2019).
- Akaike, T., Ida, T., Wei, F.Y., Nishida, M., Kumagai, Y., Alam, M.M., Ihara, H., Sawa, T., Matsunaga, T., Kasamatsu, S., Nishimura, A., Morita, M., Tomizawa, K., Nishimura, A., Watanabe, S., Inaba, K., Shima, H., Tanuma, N., Jung, M., Fujii, S., Watanabe, Y., Ohmuraya, M., Nagy, P., Feelisch, M., Fukuto, J.M. & Motohashi, H. Cysteinyl-tRNA synthetase governs cysteine polysulfidation and mitochondrial bioenergetics. Nat. Commun. 8, 1177 (2017).
A01: 代謝アダプテーション
がん微小環境における代謝適応フィードバックシステムの解明
 研究代表者:大澤 毅(東京大学・先端科学技術研究センター・特任准教授)
研究代表者:大澤 毅(東京大学・先端科学技術研究センター・特任准教授)
HPhttps://www.onc.rcast.u-tokyo.ac.jp/ja/
概要
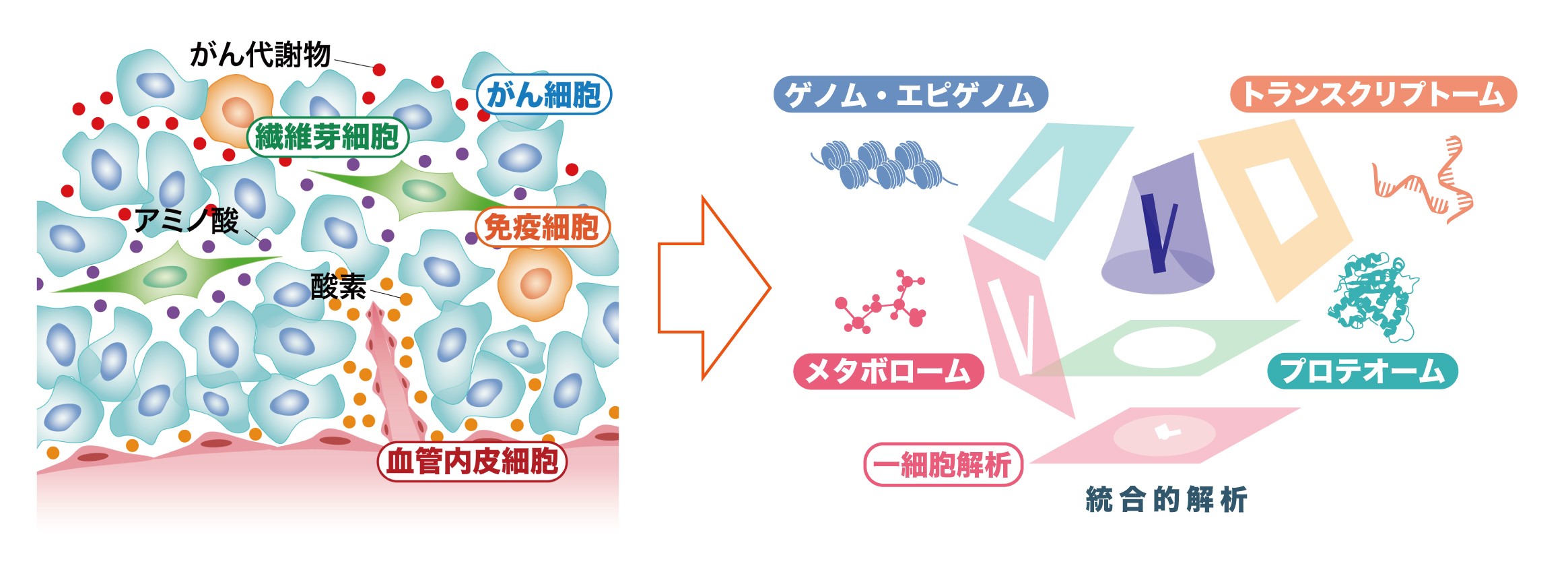
がんの浸潤・転移・薬剤耐性などの悪性化には過酷な腫瘍微小環境に適応する多重のがん代謝適応システム、すなわち「代謝アダプテーション」が重要な役割を果たす。申請者は、これまでゲノム・エピゲノム・トランスクリプトーム・メタボロームの多階層オミクスの統合的な解析から、「腫瘍微小環境」という過酷な環境変化に適応して、がん悪性化や治療抵抗性を促進することを発見してきた。具体的には、申請者らは、① エピゲノム因子ががん悪性化を促進すること、② 低pHでSREBP2が酢酸代謝を制御しがんの進展に関与すること、③ 低栄養で誘導される長鎖非コードRNA(LncRNA)ががんの悪性化を促進すること、④新規がん代謝物(オンコメタボライト)が栄養飢餓耐性に寄与すること、を見出し報告してきた。
本研究は、平成30〜令和元年度の新学術「代謝統合オミクス」研究で培った、腫瘍微小環境におけるトランスオミクス解析をさらに発展させ、「代謝アダプテーション」の視点から、がん悪性化の本質となる「がん代謝物による代謝フィードバックシステム」の包括的理解と攻略を目指し、新たながん治療法の開発につなげる。
参考文献
- Osawa, T., Shimamura, T., Saito, K., Hasegawa, Y., Ishii, N., Nishida, M., Ando, R., Kondo, A., Anwar, M., Tsuchida, R., Hino, S., Sakamoto, A., Igarashi, K., Saitoh, K., Kato, K., Endo, K., Yamano, S., Kanki, Y., Matsumura, Y., Minami, T., Tanaka, T., Anai, M., Wada, Y., Wanibuchi, H., Hayashi, M., Hamada, A., Yoshida, M., Yachida, S., Nakao, M., Sakai, J., Aburatani, H., Shibuya, M., Hanada, K., Miyano, S., Soga, T., Kodama, T. Phosphoethanolamine Accumulation Protects Cancer Cells under Glutamine Starvation through Downregulation of PCYT2, Cell Rep. 29, 89-103, (2019).
- Kondo, A., Nonaka, A., Shimamura, T., Yamamoto, S., Yoshida, T., Kodama, T., Aburatani, H., and Osawa, T. Long Noncoding RNA JHDM1D-AS1 Promotes Tumor Growth by Regulating Angiogenesis in Response to Nutrient Starvation. Mol. Cell. Biol. 37, e00125-17, (2017).
- Kondo, A., Yamamoto, S., Nakaki, R., Shimamura, T., Hamakubo, T., Sakai, J., Kodama, T., Yoshida, T., Aburatani, H., and Osawa, T. Extracellular Acidic pH Activates the Sterol Regulatory Element-Binding Protein 2 to Promote Tumor Progression. Cell Rep. 18, 2228-2242, (2017).
A01: 代謝アダプテーション
代謝入力による脂肪細胞の遺伝子発現機構の解明
 研究代表者:酒井 寿郎(東京大学・先端科学技術研究センター・教授)
研究代表者:酒井 寿郎(東京大学・先端科学技術研究センター・教授)
HPhttp://www.mm.rcast.u-tokyo.ac.jp/
概要
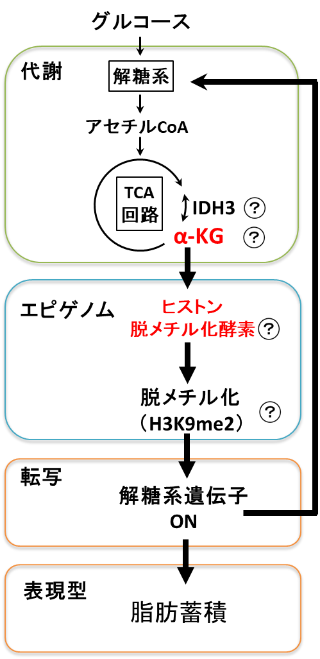 生活習慣病の発症に脂肪細胞の機能破綻が原因で発症するメカニズムが注目され、脂肪細胞の分化と機能制御メカニズムの解明は重要な課題となってきている。しかし、変化する環境にどのように前駆細胞は運命を決定し、成熟脂肪細胞に分化し環境に適応していくのか、代謝細胞運命(分化・増殖・遊走)決定のメカニズムは明らかにされていない。 本研究では環境からの代謝シグナルがどのようにして遺伝子発現そして、細胞の機能を変化させるのか否か解析し、そしてその詳細な機構を明らかにする。
生活習慣病の発症に脂肪細胞の機能破綻が原因で発症するメカニズムが注目され、脂肪細胞の分化と機能制御メカニズムの解明は重要な課題となってきている。しかし、変化する環境にどのように前駆細胞は運命を決定し、成熟脂肪細胞に分化し環境に適応していくのか、代謝細胞運命(分化・増殖・遊走)決定のメカニズムは明らかにされていない。 本研究では環境からの代謝シグナルがどのようにして遺伝子発現そして、細胞の機能を変化させるのか否か解析し、そしてその詳細な機構を明らかにする。
本研究では環境からの代謝シグナルがどのようにして遺伝子発現そして、細胞の機能を変化させるのか否か解析し、そしてその詳細な機構を明らかにする。このために、エピゲノム修飾によるクロマチン構造を変化させる分子基盤を解明し、代謝変化による脂肪細胞の分化制御機構の解明を目的とする。具体的には、① 脂肪蓄積(表現型)、② 遺伝子発現、③ メタボローム、④ エピゲノムの統合的オミクス解析をおこなう。そして脂肪細胞が細胞外の栄養を感知し、それを核内にエピゲノム変化として伝えることで、糖を取り込みやすい、脂肪を蓄積しやすい特徴を獲得することを示す。
参考文献
- Abe, Y., Fujiwara, Y., Takahashi, H., ... Sakai, J. Histone demethylase JMJD1A coordinates acute and chronic adaptation to cold stress via thermogenic phospho-switch. Nat. Commun. 9, 1566 (2018).
- Matsumura, Y., Nakaki, R., Inagaki, T., Yoshida, A., Kano, Y., Kimura, H., ... Sakai, J. H3K4/H3K9me3 bivalent chromatin domains targeted by lineage-specific DNA methylation pauses adipocyte differentiation. Molecular cell 60, 584-596 (2015).
- Abe, Y., Rozqie, R., Matsumura, Y., Kawamura, T., Nakaki, R., Tsurutani, Y., ... Sakai, J. JMJD1A is a signal-sensing scaffold that regulates acute chromatin dynamics via SWI/SNF association for thermogenesis. Nat. Commun. 6, 7052 (2015).
A01: 代謝アダプテーション
メタボロミクス可視化センサーによるニューロンの代謝調節機能の解明
 研究代表者:坪井 貴司(東京大学・大学院総合文化研究科・教授)
研究代表者:坪井 貴司(東京大学・大学院総合文化研究科・教授)
HPhttps://lci.c.u-tokyo.ac.jp/
概要
 ヒトにおいて、体重の約2%の重さの脳が、1日に消費するエネルギーの約20%を消費する。脳を構成するニューロンはエネルギー源として、グリア細胞の一種であるアストロサイトからグルコースだけを受け取るとこれまで考えられていた。しかし、ニューロンは乳酸をアストロサイトから受け取るという、「アストロサイトーニューロン乳酸経路(astrocyte-neuron lactate shuttle: ANLS)仮説」が提唱されたが、現在までにその仮説の検証には至っていない。その要因として、アストロサイトおよびニューロンにおいてグルコース代謝に関わる分子群の動態をリアルタイムで可視化解析できる技術が存在しないことが挙げられる。そこで本研究では、ANLSに関与するグルコース、乳酸、ピルビン酸、ATPを可視化するための蛍光タンパク質プローブを開発する。そして、開発したプローブを用いて、アストロサイト―ニューロン間でのこれら分子群の動態を可視化解析することで、ANLS仮説の検証を行う。
ヒトにおいて、体重の約2%の重さの脳が、1日に消費するエネルギーの約20%を消費する。脳を構成するニューロンはエネルギー源として、グリア細胞の一種であるアストロサイトからグルコースだけを受け取るとこれまで考えられていた。しかし、ニューロンは乳酸をアストロサイトから受け取るという、「アストロサイトーニューロン乳酸経路(astrocyte-neuron lactate shuttle: ANLS)仮説」が提唱されたが、現在までにその仮説の検証には至っていない。その要因として、アストロサイトおよびニューロンにおいてグルコース代謝に関わる分子群の動態をリアルタイムで可視化解析できる技術が存在しないことが挙げられる。そこで本研究では、ANLSに関与するグルコース、乳酸、ピルビン酸、ATPを可視化するための蛍光タンパク質プローブを開発する。そして、開発したプローブを用いて、アストロサイト―ニューロン間でのこれら分子群の動態を可視化解析することで、ANLS仮説の検証を行う。
参考文献
- Nakamura T, Harada K, Kamiya T, Takizawa M, Kuppers J, Nakajima K, Gutschow M, Kitaguchi T, Ohta K, Kato T, Tsuboi T. Glutamine-induced signaling pathways via amino acid receptors in enteroendocrine L cell line. Journal of Molecular Endocrinology 64, 133-143 (2020)
- Mita, M., Ito, M., Harada, K., Sugawara, I., Ueda, H., Tsuboi, T., & Kitaguchi, T. Green fluoresecent protein-based glucose indicators report glucose dynamics in living cells. Analytical Chemistry 91, 4821-4830 (2019).
- Arai, S., Kriszt, R., Harada, K., Looi, LS, Matsuda, S., Wongso, D., Suo, S., Ishiura, S., Tseng, YH., Raghunath, M., Ito, T., Tsuboi, T., Kitaguchi, T. RGB-color intensiometric indicators visualize spatiotemporal dynamics of ATP in single cells. Angewandte Chemie International Edition 57, 10873-10878 (2018).
A01: 代謝アダプテーション
転写後遺伝子発現制御に着目した低酸素応答の代謝アダプテーションの機序解明

| 研究代表者: | 秋光 信佳(東京大学・アイソトープ総合センター・教授) |
| 研究協力者: | 川村 猛(東京大学・アイソトープ総合センター・准教授) 川田 健太郎(東京大学・アイソトープ総合センター・特任助教) |
HPhttps://akimitsu.ric.u-tokyo.ac.jp/
概要
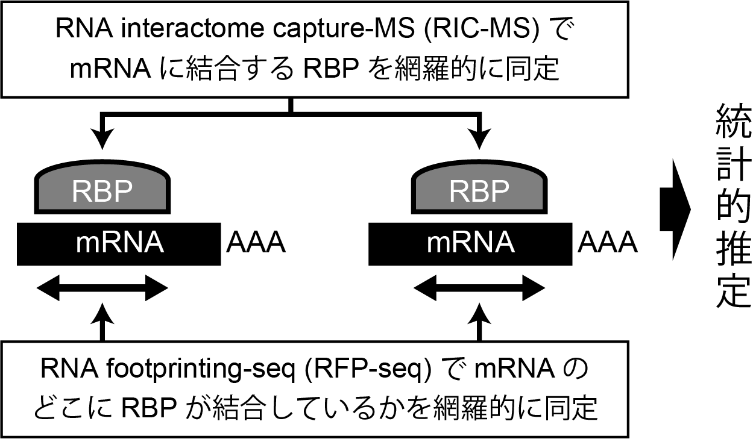 遺伝子発現量は、転写因子による転写調節とRNA結合タンパク質(RBP)による転写後調節(RNA分解調節等)のバランスで決定される。転写後調節の詳細な機構を解明するためには、それぞれのmRNAに結合するRBPを網羅的に同定することが重要である。すなわち、多対多の相互作用の全貌解明が重要である。しかしながら、RBPの配列認識の特異性が低いことが多対多の相互作用解明を困難にしている。本研究ではmRNAに結合するRBPを網羅的に同定する新規手法Transcriptome-RBPome Interaction Analysis pipeLine (TRIAL) を開発する。本研究は以下の3つの段階から成る。(1)初めに、mRNAに結合しているRBPを網羅的に同定するRNA interactome capture-MS(RIC-MS)を確立する。なおこの段階では、RBPがどのmRNAに結合するかは不明である。(2)次に、mRNAのどこにRBPが結合するかを網羅的に同定するRNA footprinting-sequencing(RFP-seq)を確立する。なおこの段階では、mRNAに結合するRBPの種類は不明である。(3)最後に、RBPおよびmRNAの分布を比較することで、それぞれのmRNAに結合するRBPを統計的に推定する。最終的にこれらの開発を通して、低酸素に応答した代謝アダプテーションの詳細な機構を転写後制御(RNA分解制御)の観点から解明する。
遺伝子発現量は、転写因子による転写調節とRNA結合タンパク質(RBP)による転写後調節(RNA分解調節等)のバランスで決定される。転写後調節の詳細な機構を解明するためには、それぞれのmRNAに結合するRBPを網羅的に同定することが重要である。すなわち、多対多の相互作用の全貌解明が重要である。しかしながら、RBPの配列認識の特異性が低いことが多対多の相互作用解明を困難にしている。本研究ではmRNAに結合するRBPを網羅的に同定する新規手法Transcriptome-RBPome Interaction Analysis pipeLine (TRIAL) を開発する。本研究は以下の3つの段階から成る。(1)初めに、mRNAに結合しているRBPを網羅的に同定するRNA interactome capture-MS(RIC-MS)を確立する。なおこの段階では、RBPがどのmRNAに結合するかは不明である。(2)次に、mRNAのどこにRBPが結合するかを網羅的に同定するRNA footprinting-sequencing(RFP-seq)を確立する。なおこの段階では、mRNAに結合するRBPの種類は不明である。(3)最後に、RBPおよびmRNAの分布を比較することで、それぞれのmRNAに結合するRBPを統計的に推定する。最終的にこれらの開発を通して、低酸素に応答した代謝アダプテーションの詳細な機構を転写後制御(RNA分解制御)の観点から解明する。
参考文献
- Yamada T., Imamachi N., Imamura K., Taniue K., Kawamura T., Suzuki Y., Nagahama M., Akimitsu N. (2020) Systematic Analysis of Targets of Pumilio-mediated mRNA Decay Reveals that PUM1 Repression by DNA Damage Activates Translesion Synthesis, Cell Rep. 31, 107542.
- Imamachi N., Salam K.A., Suzuki Y. and Akimitsu N. (2017) A GC-rich sequence feature in the 3’ UTR directs UPF1-dependent mRNA decay in mammalian cells. Genome Res. 27, 407-418.
- Tani H., Mizutani R., Salam KA. Tano K., Ijiri K., Wakamatsu A., Isogai T., Suzuki Y. and Akimitsu N. (2012) Genome-wide determination of RNA stability reveals hundreds of short-lived non-coding transcripts in mammals, Genome Res., 22, 947-956.
A01: 代謝アダプテーション
がんに起因する多臓器代謝異常に対する宿主アダプテーションのトランスオミクス解析
 研究代表者:河岡 慎平(京都大学 ウイルス・再生医科学研究所 特定准教授)
研究代表者:河岡 慎平(京都大学 ウイルス・再生医科学研究所 特定准教授)
HPhttps://scholar.google.com/citations?user=jDL3tGUAAAAJ&hl=en
概要
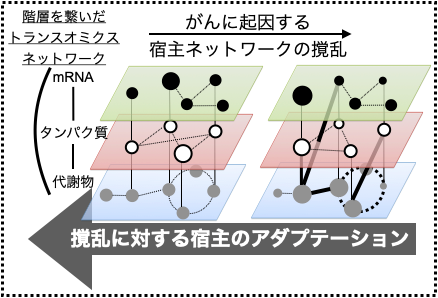 あらゆるがんを根治することは人類の悲願ではあるが、根治不能な症例の数は依然として多い。たとえば我が国では、年間少なくとも37万人が、根治不能ながんを抱えたままの生活を余儀なくされている 。根治不能ながんは脂肪や肝臓、筋肉などの代謝臓器にさまざまな悪影響を与え、全身性の代謝障害をひきおこす。この代謝障害は、臨床的にはがん悪液質 (がんカヘキシア) として知られる。しかし、がんが根治不能と判断されるような場合であっても、代謝障害の全てがすぐに顕在化し、生命に危険が及ぶわけではない。このことは、宿主が、がんによる撹乱に適応 (アダプテーション) し、生命を維持するしくみを備えていることを示唆する。がんによる撹乱への宿主のアダプテーションは、がんと宿主が関与しあう複雑な現象であり、その全体像は未解明である。本公募班では、担がんマウスモデルと多階層オミクス、そしてマウス遺伝学 (エンハンサー遺伝学を含む) をくみあわせて、がんに起因する多臓器の代謝異常に対する宿主のアダプテーションの実態を明らかにする。本領域の研究環境を活かして、がんに影響される宿主臓器どうしの関係性 (臓器連関) についても明らかにしたい。
あらゆるがんを根治することは人類の悲願ではあるが、根治不能な症例の数は依然として多い。たとえば我が国では、年間少なくとも37万人が、根治不能ながんを抱えたままの生活を余儀なくされている 。根治不能ながんは脂肪や肝臓、筋肉などの代謝臓器にさまざまな悪影響を与え、全身性の代謝障害をひきおこす。この代謝障害は、臨床的にはがん悪液質 (がんカヘキシア) として知られる。しかし、がんが根治不能と判断されるような場合であっても、代謝障害の全てがすぐに顕在化し、生命に危険が及ぶわけではない。このことは、宿主が、がんによる撹乱に適応 (アダプテーション) し、生命を維持するしくみを備えていることを示唆する。がんによる撹乱への宿主のアダプテーションは、がんと宿主が関与しあう複雑な現象であり、その全体像は未解明である。本公募班では、担がんマウスモデルと多階層オミクス、そしてマウス遺伝学 (エンハンサー遺伝学を含む) をくみあわせて、がんに起因する多臓器の代謝異常に対する宿主のアダプテーションの実態を明らかにする。本領域の研究環境を活かして、がんに影響される宿主臓器どうしの関係性 (臓器連関) についても明らかにしたい。
参考文献
- Hojo MA, Masuda K, Hojo H, Nagahata Y, Yasuda K, Ohara D, Takeuchi Y, Hirota K, Suzuki Y Kawamoto H, and Kawaoka S. Identification of a genomic enhancer that enforces proper apoptosis induction in thymic negative selection. Nature Communications 2019; 10: 2603.
- Enya S, Kawakami K, Suzuki Y, and Kawaoka S. A novel zebrafish intestinal tumor model reveals a role for cyp7a1-dependent tumor-liver crosstalk in causing adverse effects on the host. Disease Models & Mechanisms 2018 11(8). pii: dmm032383.
- Hojo H, Enya S, Arai M, Suzuki Y, Nojiri T, Kangawa K, Koyama S and Kawaoka S. Remote reprogramming of hepatic circadian transcriptome by breast cancer. Oncotarget 2017. 8:34128-34140
A01: 代謝アダプテーション
冬眠・人工冬眠・生命保護を結ぶ代謝アダプテーション
 研究代表者:小早川 令子(関西医科大学・医学部・教授)
研究代表者:小早川 令子(関西医科大学・医学部・教授)
概要
 (工事中)
(工事中)
参考文献
- (工事中)
A01: 代謝アダプテーション
乾燥ストレスに対する植物の代謝アダプテーション機構の解明
 研究代表者:平井 優美(理化学研究所・環境資源科学研究センター・チームリーダー)
研究代表者:平井 優美(理化学研究所・環境資源科学研究センター・チームリーダー)
HPhttp://www.csrs.riken.jp/jp/labs/msrt/
概要
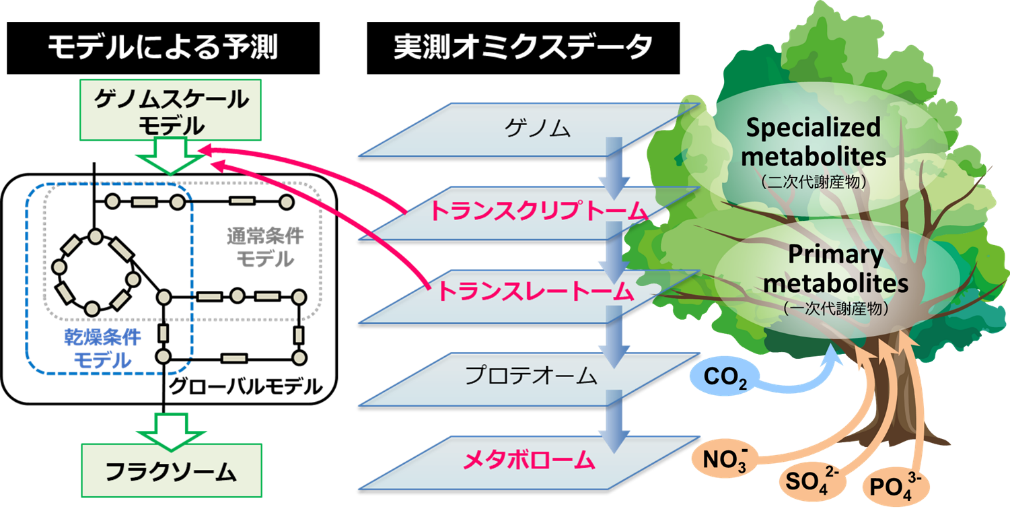 地面に根を下ろして動かない植物は静的なイメージがあるが、環境変化に応答、適応して生き延びる生存戦略を取っているためむしろ動物よりダイナミックな代謝アダプテーションの機構を有していると考えられる。炭素のフラックスだけに着目しても、光合成によりCO2を同化して作った糖を、成長段階や環境変動に応じてバイオマス、エネルギー、貯蔵、二次代謝へと自在に分配する精密な制御機構を植物が有していることがうかがえる。本研究では、乾燥ストレスに対する植物の代謝アダプテーションのメカニズムを、トランスオミクス解析によって解明することを目指す。シロイヌナズナのゲノムスケール代謝モデルを基に、乾燥条件下で取得するトランスクリプトームおよびトランスレートームデータの利用によって乾燥条件固有モデルを構築して、乾燥条件下でのフラックス分布を推定する。また、乾燥条件下で転写産物量または翻訳量の制御によってフラックスが決定される代謝反応を明らかにするとともに、乾燥ストレスへの代謝アダプテーションを実現する鍵となる代謝反応を同定する。
地面に根を下ろして動かない植物は静的なイメージがあるが、環境変化に応答、適応して生き延びる生存戦略を取っているためむしろ動物よりダイナミックな代謝アダプテーションの機構を有していると考えられる。炭素のフラックスだけに着目しても、光合成によりCO2を同化して作った糖を、成長段階や環境変動に応じてバイオマス、エネルギー、貯蔵、二次代謝へと自在に分配する精密な制御機構を植物が有していることがうかがえる。本研究では、乾燥ストレスに対する植物の代謝アダプテーションのメカニズムを、トランスオミクス解析によって解明することを目指す。シロイヌナズナのゲノムスケール代謝モデルを基に、乾燥条件下で取得するトランスクリプトームおよびトランスレートームデータの利用によって乾燥条件固有モデルを構築して、乾燥条件下でのフラックス分布を推定する。また、乾燥条件下で転写産物量または翻訳量の制御によってフラックスが決定される代謝反応を明らかにするとともに、乾燥ストレスへの代謝アダプテーションを実現する鍵となる代謝反応を同定する。
参考文献
- Siriwach, R., Matsuda, F., Yano, K. & Hirai, M.Y. Drought stress responses in context-specific genome-scale metabolic models of Arabidopsis thaliana. Metabolites 10, 159 (2020).
- Hirai, M.Y. & Shiraishi, F. Using metabolome data for mathematical modeling of plant metabolic systems. Current Opinion in Biotechnoogy 54, 138-144 (2018).
- Sriyudthsak, K., Mejia, R.F., Arita, M. & Hirai, M.Y. PASMet: a web-based platform for prediction, modelling and analyses of metabolic systems. Nucleic Acids Research 44, W205-W211 (2016).
A01: 代謝アダプテーション
線虫の幼虫期発生プログラムを起動する栄養シグナルと遺伝子発現ネットワークの解明
 研究代表者:黒柳 秀人(琉球大学・大学院医学研究科・教授)
研究代表者:黒柳 秀人(琉球大学・大学院医学研究科・教授)
HPhttp://biochem.med.u-ryukyu.ac.jp/
概要
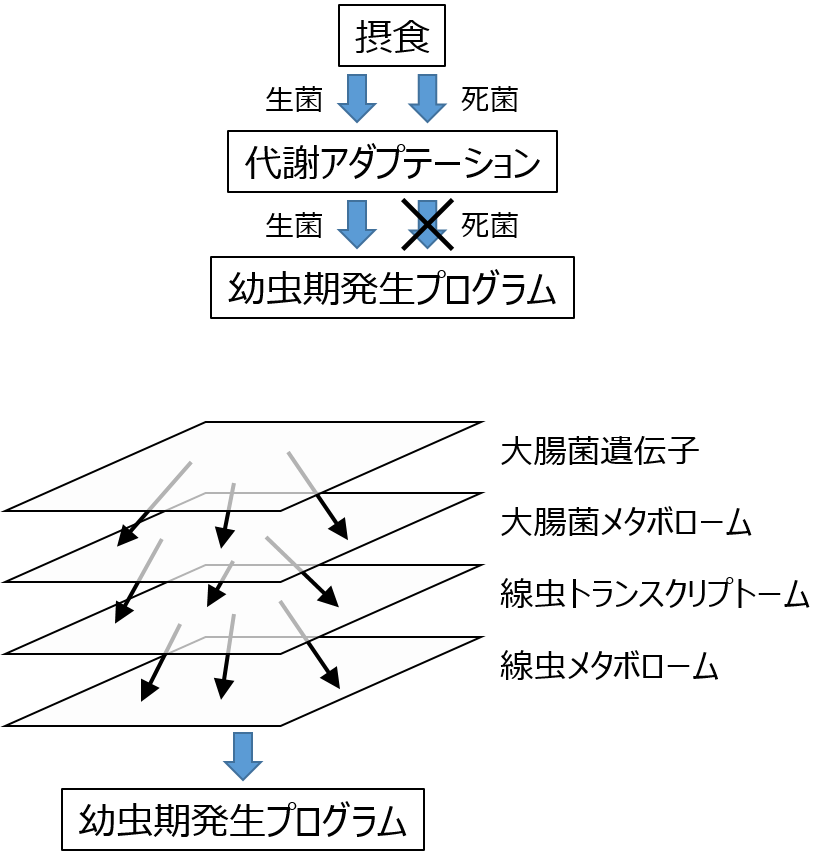 モデル多細胞生物である線虫Caenorhabditis elegansの孵化したばかりのL1期幼虫にとって、摂食は幼虫発生期へ進むための重要な外界シグナルである。線虫は実験室で大腸菌を食餌として飼育されているが、近年、線虫と大腸菌の関係が単なる捕食者と食餌の関係から、代謝や寿命に影響を与える共生者としての面が認識されるようになっている。我々は、大腸菌を溶菌した溶解産物では線虫が成長できないにも拘わらず、代謝酵素遺伝子などの発現/抑制や選択的スプライシングなどの遺伝子発現応答が生大腸菌と同程度に大きく誘導されることを見出した。しかし、線虫が幼虫期発生プログラムを開始するために食餌が生菌である必要性の根拠は今日でも具体的には明らかにされていない。
モデル多細胞生物である線虫Caenorhabditis elegansの孵化したばかりのL1期幼虫にとって、摂食は幼虫発生期へ進むための重要な外界シグナルである。線虫は実験室で大腸菌を食餌として飼育されているが、近年、線虫と大腸菌の関係が単なる捕食者と食餌の関係から、代謝や寿命に影響を与える共生者としての面が認識されるようになっている。我々は、大腸菌を溶菌した溶解産物では線虫が成長できないにも拘わらず、代謝酵素遺伝子などの発現/抑制や選択的スプライシングなどの遺伝子発現応答が生大腸菌と同程度に大きく誘導されることを見出した。しかし、線虫が幼虫期発生プログラムを開始するために食餌が生菌である必要性の根拠は今日でも具体的には明らかにされていない。
本研究では、線虫のL1期幼虫が様々な種類の食餌の摂食により代謝酵素の遺伝子発現を柔軟に変動させて体内代謝環境を整え、幼虫期発生プログラムを開始する運命決定に至る過程を代謝アダプテーションとして捉える。食餌が生菌の場合と死菌の場合で共通/相違する線虫の遺伝子発現応答をRNA-seq解析で明らかにすることで、生菌に特異的で幼虫期発生プログラムの開始に必須の遺伝子発現の時期と制御経路を特定する。さらに、食餌である生菌と死菌あるいは大腸菌と他の細菌のメタボローム解析と、摂食前および摂食後に幼虫期発生プログラムの開始可否を決定する時点での線虫のメタボローム解析を組み合わせることで、摂食に対する遺伝子発現応答による代謝アダプテーションの全体像を明らかにする。最終的な目標として、幼虫期発生開始に必須の生菌中の代謝産物の特定を目指す。
参考文献
- Ihara K, Sasano T, Hiraoka Y, Togo-Ohno M, Soejima Y, Sawabe M, Tsuchiya M, Ogawa H, Furukawa T, Kuroyanagi H. A missense mutation in the RSRSP stretch of Rbm20 causes dilated cardiomyopathy and atrial fibrillation in mice. Scientific Reports 10, 17894 (2020).
- Arribere, J.A., Kuroyanagi, H., Hundley, H.A. mRNA Editing, Processing and Quality Control in Caenorhabditis elegans. Genetics 215, 531-568 (2020).
- Barnes, D.E., Watabe, E., Ono, K., Kwak, E., Kuroyanagi, H., Ono, S. Tropomyosin isoforms differentially affect muscle contractility in the head and body regions of the nematode Caenorhabditis elegans. Molecular Biology of the Cell 29, 1075-1088 (2018).
A02: トランスオミクス解析技術開発
臓器連関トランスオミクスを読み解くベイズモデリング技術の開発
 研究代表者:島村 徹平(名古屋大学・大学院医学系研究科・教授)
研究代表者:島村 徹平(名古屋大学・大学院医学系研究科・教授)
研究協力者:河岡 慎平(京都大学・ウイルス・再生医科学研究所・特定准教授)
HPhttp://www.nagoya-sysbiol.info/
概要
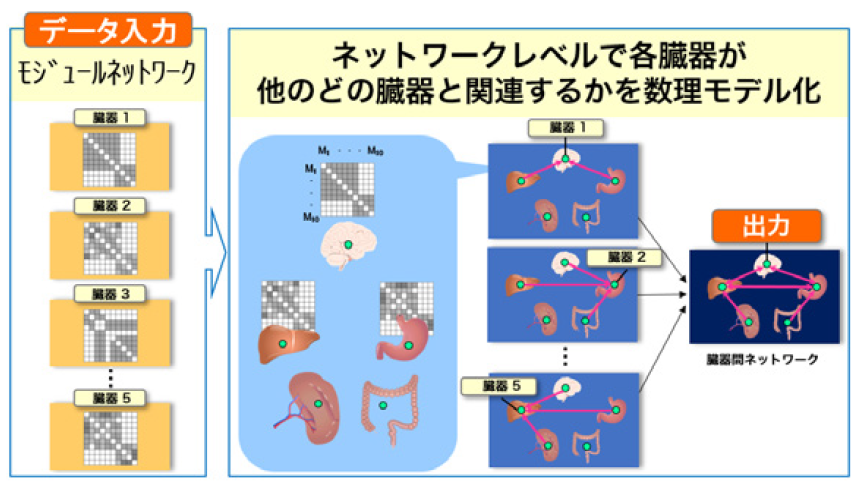 細胞内代謝アダプテーションの分子機序を理解し、階層間のクロストークを解明するためには、生命現象を階層ごとの閉じた情報で捉える従前のアプローチから脱却し、各階層の情報を統合し、階層をまたがる生体分子間の相互作用ネットワークを記述するデータ駆動型の数理モデルの開発が必要である。本研究では、代謝アダプテーションの臓器横断的理解を加速する下記の情報解析技術として、(1)臓器横断的モジュールを同定するためのベイズモデリング技術、(2)臓器間、臓器内相互作用を解明するための機械学習技術を開発する。これら解析技術の有用性を、ヒト臨床サンプル、疾患モデル動物を用いた実験系で検証する。本研究の推進により、生命情報を階層別に解析することで生じる情報損失を回避するとともに、分子、細胞、臓器という異なるスケールの情報を繋ぎ、階層間・臓器関の因果律に迫るための情報解析基盤の構築、複雑な生命現象をマルチオミクスとマルチスケールの観点から俯瞰的に思考する新しいパラダイムの創出を目指す。
細胞内代謝アダプテーションの分子機序を理解し、階層間のクロストークを解明するためには、生命現象を階層ごとの閉じた情報で捉える従前のアプローチから脱却し、各階層の情報を統合し、階層をまたがる生体分子間の相互作用ネットワークを記述するデータ駆動型の数理モデルの開発が必要である。本研究では、代謝アダプテーションの臓器横断的理解を加速する下記の情報解析技術として、(1)臓器横断的モジュールを同定するためのベイズモデリング技術、(2)臓器間、臓器内相互作用を解明するための機械学習技術を開発する。これら解析技術の有用性を、ヒト臨床サンプル、疾患モデル動物を用いた実験系で検証する。本研究の推進により、生命情報を階層別に解析することで生じる情報損失を回避するとともに、分子、細胞、臓器という異なるスケールの情報を繋ぎ、階層間・臓器関の因果律に迫るための情報解析基盤の構築、複雑な生命現象をマルチオミクスとマルチスケールの観点から俯瞰的に思考する新しいパラダイムの創出を目指す。
参考文献
- Abe K, Hirayama M, Ohno K, Shimamura T. ENIGMA: an enterotype-like unigram mixture model for microbial association analysis. BMC Genomics 20(Suppl 2):191 (2019).
- Osawa T, Shimamura T, Saito K, Hasegawa Y, Ishii N, Nishida M, Ando R, Kondo A, Anwar M, Tsuchida R, Hino S, Sakamoto A, Igarashi K, Saitoh K, Kato K, Endo K, Yamano S, Kanki Y, Matsumura Y, Minami T, Tanaka T, Anai M, Wada Y, Wanibuchi H, Hayashi M, Hamada A, Yoshida M, Yachida S, Nakao M, Sakai J, Aburatani H, Shibuya M, Hanada K, Miyano S, Soga T, Kodama T. Phosphoethanolamine Accumulation Protects Cancer Cells under Glutamine Starvation through Downregulation of PCYT2. Cell Rep 29(1):89-103.e7 (2019).
- Kawakubo H, Matsui Y, Kushima I, Ozaki N, Shimamura T. A network of networks approach for modeling interconnected brain tissue-specific networks. Bioinformatics 35(17):3092-3101 (2019).
A02: トランスオミクス解析技術開発
摂動による迅速な翻訳応答を捉えるプロテオームリボソームプロファイリング法の開発

| 研究代表者: | 今見 考志(京都大学・大学院薬学研究科・特任講師) |
| 研究協力者: | 小形 公亮(同・特定助教) 石濱 泰(同・教授) |
HPhttp://www.pharm.kyoto-u.ac.jp/seizai/
概要
細胞は摂動や環境によって転写プロファイルや(リン酸化)プロテオームプロファイルを変化させ環境に適用する。申請者の最近の研究(参考文献参照)も含め、転写を介さずに転写後・翻訳レベルで迅速にプロテオームを変化させることで細胞機能を制御している例が報告されている。本研究では、質量分析装置を用いて、リボソーム上で翻訳された直後のペプチド配列を大規模にプロファイルするテクノロジーを創出する。さらに本法を用いて、従来の総タンパク質を対象としたプロテオーム解析では捉えることが困難であった摂動によって誘導される分オーダーの翻訳応答をモニターし、翻訳レベルでの迅速なタンパク質合成制御機構を明らかにする。
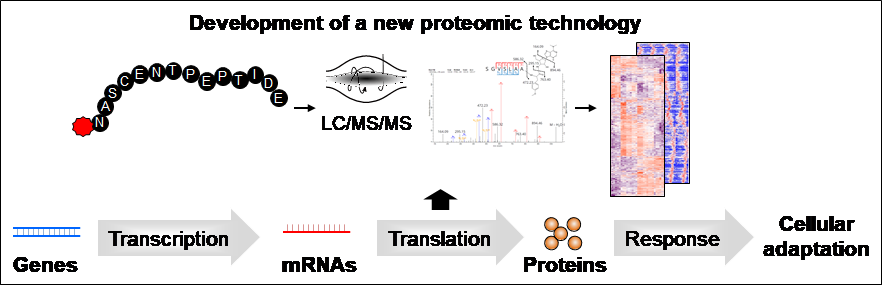
参考文献
- Imami K# Milek M, Bogdanow B, Yasuda Y, Kastelic N, Zauber H, Ishihama Y, Landthaler M, Selbach M# (#corresponding author) Phosphorylation of the ribosomal protein RPL12/uL11 affects translation during mitosis. Molecular Cell, 4;72(1):84-98 (2018).
- Imami K#, Yasuda T Measuring protein synthesis during cell cycle by azidohomoalanine (AHA) labeling and flow cytometric analysis Bio-Protocol, Vol 9, Issue 8 (2019).
- Wessel H*, Imami K*, Baltz A, Kolinksi M, Beldovskaya A, Selbach M, Small S, Ohler U, Landthaler M (*co-first author) The mRNA-bound proteome of the early fly embryo. Genome Research, 26: pp1000-1009 (2016).
A02: トランスオミクス解析技術開発
活性制御部位関連ペプチドによるキノーム活性測定法の開発
 研究代表者:杉山 直幸(京都大学・大学院薬学研究科・准教授)
研究代表者:杉山 直幸(京都大学・大学院薬学研究科・准教授)
HPhttp://www.pharm.kyoto-u.ac.jp/seizai/
概要
タンパク質リン酸化酵素(プロテインキナーゼ)の活性制御部位について、その部位を含むリン酸化ペプチドを高感度に検出する手法を開発し、代謝制御における各キナーゼ活性を評価することを目的とする。
ヒトでは、500種類以上のタンパク質リン酸化酵素(プロテインキナーゼ)が存在しており、代謝制御においても重要な役割を果たしていると考えられるが、その機能については十分明らかにされていない。研究代表者らはこれまでの研究により、385種類のヒトキナーゼのin vitro基質の同定とリン酸化モチーフを抽出している。さらに、摂動を与えた細胞中におけるタンパク質の大規模リン酸化情報(リン酸化プロテオーム)と人工基質ペプチドライブラリを用いた試料中の全キナーゼ(キノーム)の活性計測を行うことで、キナーゼの活性制御部位の同定を行った。
本研究では人工基質ペプチドの再設計による対象キナーゼの拡大を行い、取得した各キナーゼの活性制御部位を含むペプチドを測定対象として、高選択的リン酸化ペプチド濃縮法とターゲットプロテオミクスを組み合わせることでキナーゼ活性をキノーム(全キナーゼ)レベルで定量することを目指す。
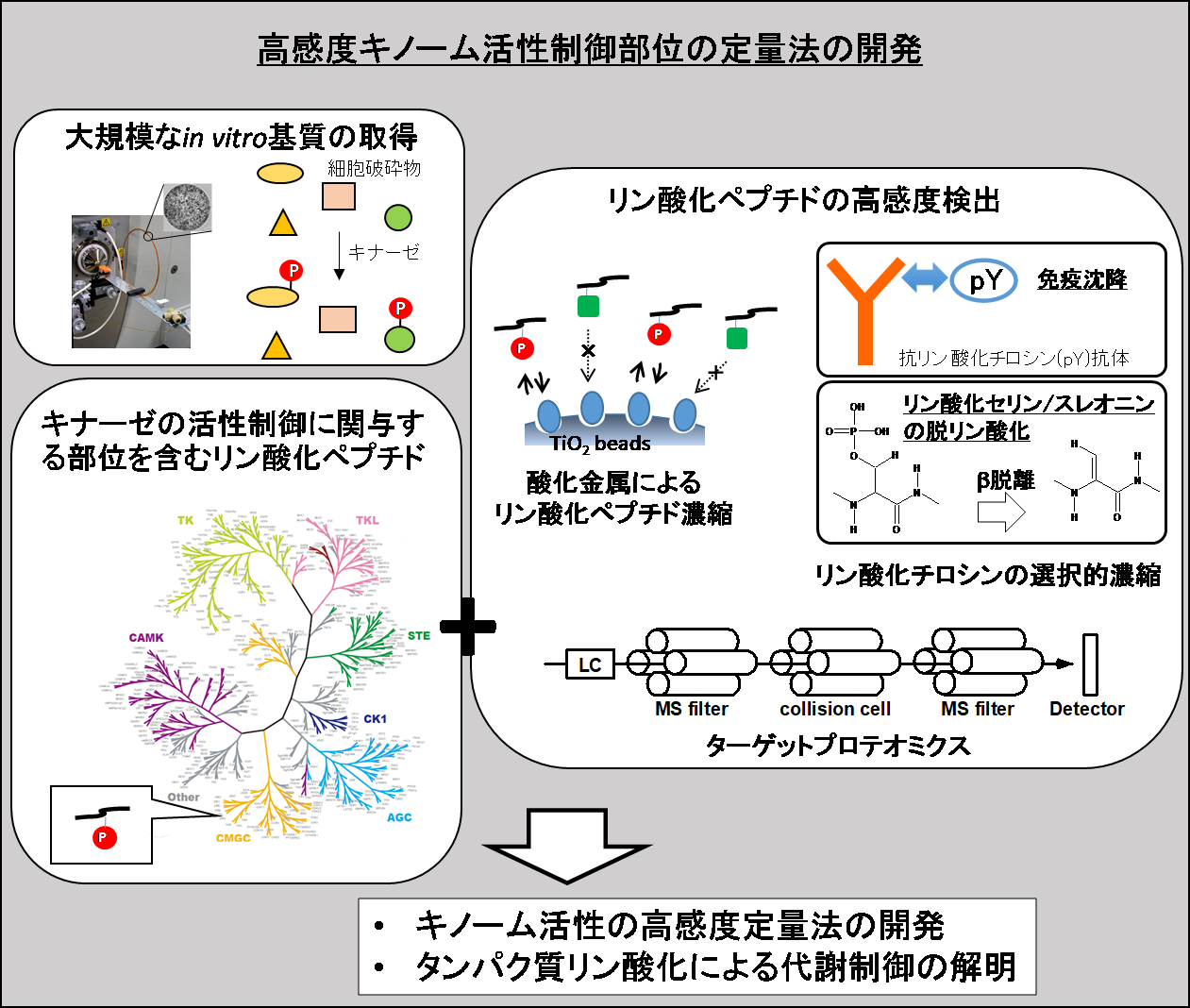
参考文献
- Sugiyama N, Imamura H & Ishihama Y. Large-scale Discovery of Substrates of the Human Kinome. Sci Rep. 9, 10503. (2019).
- Takahashi C, Yazaki T, Sugiyama N & Ishihama Y. Selected reaction monitoring of kinase activity-targeted phosphopeptides. Chromatography 40, 39-47 (2019)
- Imamura H, Wagih O, Niinae T, Sugiyama N, Beltrao P & Ishihama Y. Identifications of Putative PKA Substrates with Quantitative Phosphoproteomics and Primary-Sequence-Based Scoring. J Proteome Res. 16, 1825-1830 (2017).
A02: トランスオミクス解析技術開発
高深度解析を可能とする単一細胞空間オミクス技術の開発
 研究代表者:大川 恭行(九州大学・生体防御医学研究所・教授)
研究代表者:大川 恭行(九州大学・生体防御医学研究所・教授)
HPhttp://tx.bioreg.kyushu-u.ac.jp/
概要
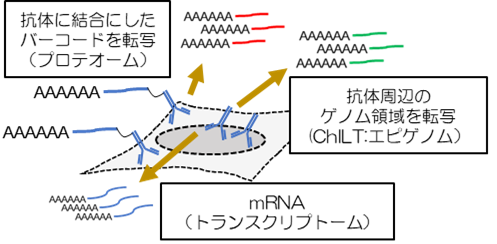 単一細胞オミクス技術は、トランスクリプトーム解析において、様々なデバイスや手法が開発され生体内の細胞を網羅的に解析する道を拓いた。単一細胞トランスクリプトーム解析の実現により、生体内情報である、ゲノム、エピゲノムからメタボロームに至るあらゆる細胞内の情報を1 細胞レベルで高深度に解析することを目標に多くの技術開発が進められている。一方で、過去1-2 年で、爆発的な進化を遂げているのは、これら“オーム”情報に組織や器官内の空間的位置情報を付加する空間オミクス技術である。現在開発されている技術の多くは、組織切片を用いたものであるが、単一細胞レベルでの網羅性に乏しく実用性に欠けている。そこで、本研究では、組織内の細胞の位置情報を保持した状態での1 細胞解像度の空間オミクス技術の開発を行う。まず空間情報を含めた単一細胞レベルでのトランスクリプトーム解析法の確立を行い、続いてプロテオーム等の他のオームの同時解析技術を樹立させる。本技術開発では、組織切片上の細胞に空間情報を識別する固有のバーコードを付加する。その後、細胞内のトランスクリプトーム、プロテオーム等の情報を人工的なRNA に転換する。そのうえで空間バーコードとRNA に置換された細胞内情報を次世代シークエンサー(NGS)の1 回のRNAseq 解析で網羅的に解析することにより、空間・位置情報に紐づけされた単
一細胞レベルの、高深度オミクス解析技術の開発を進める。
単一細胞オミクス技術は、トランスクリプトーム解析において、様々なデバイスや手法が開発され生体内の細胞を網羅的に解析する道を拓いた。単一細胞トランスクリプトーム解析の実現により、生体内情報である、ゲノム、エピゲノムからメタボロームに至るあらゆる細胞内の情報を1 細胞レベルで高深度に解析することを目標に多くの技術開発が進められている。一方で、過去1-2 年で、爆発的な進化を遂げているのは、これら“オーム”情報に組織や器官内の空間的位置情報を付加する空間オミクス技術である。現在開発されている技術の多くは、組織切片を用いたものであるが、単一細胞レベルでの網羅性に乏しく実用性に欠けている。そこで、本研究では、組織内の細胞の位置情報を保持した状態での1 細胞解像度の空間オミクス技術の開発を行う。まず空間情報を含めた単一細胞レベルでのトランスクリプトーム解析法の確立を行い、続いてプロテオーム等の他のオームの同時解析技術を樹立させる。本技術開発では、組織切片上の細胞に空間情報を識別する固有のバーコードを付加する。その後、細胞内のトランスクリプトーム、プロテオーム等の情報を人工的なRNA に転換する。そのうえで空間バーコードとRNA に置換された細胞内情報を次世代シークエンサー(NGS)の1 回のRNAseq 解析で網羅的に解析することにより、空間・位置情報に紐づけされた単
一細胞レベルの、高深度オミクス解析技術の開発を進める。
参考文献
- Harada, A., Maehara, K., Handa, T., Arimura, Y., Nogami, J., Hayashi-Takanaka, Y., ... & Ohkawa, Y. (2019). A chromatin integration labelling method enables epigenomic profiling with lower input. Nature cell biology, 21(2), 287-296.
- Harada, A., Maehara, K., Ono, Y., Taguchi, H., Yoshioka, K., Kitajima, Y., ... & Ohkawa, Y. (2018). Histone H3. 3 sub-variant H3mm7 is required for normal skeletal muscle regeneration. Nature communications, 9(1), 1-13.
- Semba, Y., Harada, A., Maehara, K., Oki, S., Meno, C., Ueda, J., ... & Ohkawa, Y. (2017). Chd2 regulates chromatin for proper gene expression toward differentiation in mouse embryonic stem cells. Nucleic acids research, 45(15), 8758-8772.
A02: トランスオミクス解析技術開発
データ駆動アプローチからのトランスオミクスネットワーク推定法の開発
 研究代表者:宇田 新介(九州大学・生体防御医学研究所・准教授)
研究代表者:宇田 新介(九州大学・生体防御医学研究所・准教授)
HPhttp://www.bioreg.kyushu-u.ac.jp/labo/omics/index.html
概要
工事中
参考文献
-
工事中
A02: トランスオミクス解析技術開発
テンソル分解を用いた教師無し学習による変数選択法を用いたトランスオミクス解析
 研究代表者:田口 善弘(中央大学・理工学部・教授)
研究代表者:田口 善弘(中央大学・理工学部・教授)
HPhttps://researchmap.jp/Yh_Taguchi/
概要
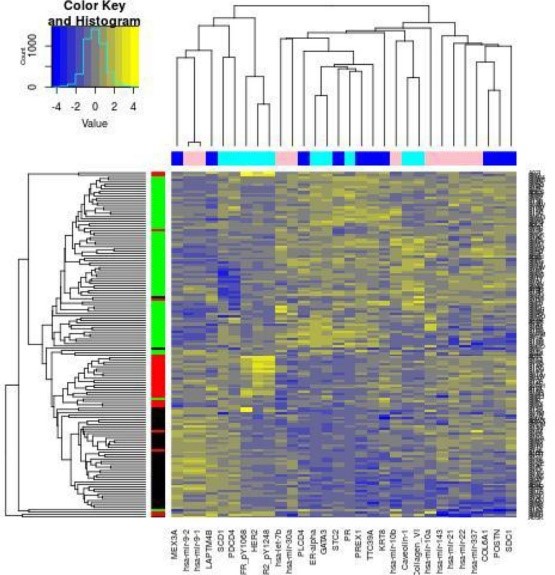 いわゆる機械学習は教師あり学習、教師無し学習、強化学習の3つに分類されるとされているが、深層学習を初めてとしてビッグデータと組み合わせて初めて性能を発揮することが多い。しかし、オミックスデータにおいては一サンプルあたりの費用が効果であり、サンプル数が稼げない一方、変数は遺伝子の総数など数万以上になることが多く、いわゆるlarge p small n問題になってしまい、効率的な機械学習の適用が難しくなっている。この困難に対して自分は「主成分分析及びテンソル分解を用いた教師無し学習による変数選択法」と提案し、2019年9月にその成果をまとめたモノグラフ(英文、単著)をシュプリンガー社から刊行した[1]。この方法はマルチオミックス解析にも有効であることが解っており[2](図は同法により選択されたmiRNA,mRNA,protein(列)が3種類のセルライン(行)をよく判別している様子を表すヒートマップである[3])、本新領域にも大きな貢献ができるものと信じる。
いわゆる機械学習は教師あり学習、教師無し学習、強化学習の3つに分類されるとされているが、深層学習を初めてとしてビッグデータと組み合わせて初めて性能を発揮することが多い。しかし、オミックスデータにおいては一サンプルあたりの費用が効果であり、サンプル数が稼げない一方、変数は遺伝子の総数など数万以上になることが多く、いわゆるlarge p small n問題になってしまい、効率的な機械学習の適用が難しくなっている。この困難に対して自分は「主成分分析及びテンソル分解を用いた教師無し学習による変数選択法」と提案し、2019年9月にその成果をまとめたモノグラフ(英文、単著)をシュプリンガー社から刊行した[1]。この方法はマルチオミックス解析にも有効であることが解っており[2](図は同法により選択されたmiRNA,mRNA,protein(列)が3種類のセルライン(行)をよく判別している様子を表すヒートマップである[3])、本新領域にも大きな貢献ができるものと信じる。
参考文献
- Taguchi, Y-h. Unsupervised Feature Extraction Applied to Bioinformatics: A PCA Based and TD Based Approach, Springer International (2019).
- Taguchi, Y-h. and Ng, K "[Regular Paper] Tensor Decomposition–Based Unsupervised Feature Extraction for Integrated Analysis of TCGA Data on MicroRNA Expression and Promoter Methylation of Genes in Ovarian Cancer," 2018 IEEE 18th International Conference on Bioinformatics and Bioengineering (BIBE), Taichung, pp. 195-200. (2018).
- Taguchi, Y-h. Multiomics Data Analysis Using Tensor Decomposition Based Unsupervised Feature Extraction. In: Huang DS., Bevilacqua V., Premaratne P. (eds) Intelligent Computing Theories and Application. ICIC 2019. Lecture Notes in Computer Science, vol 11643. Springer, Cham (2019)
A02: トランスオミクス解析技術開発
複数シグナル経路活性の同時計測を可能とする分子プローブ技術の開発
 研究代表者:足達 俊吾(産業技術総合研究所 主任研究員)
研究代表者:足達 俊吾(産業技術総合研究所 主任研究員)
HPhttps://www.molprof.jp/about/organization.html
概要
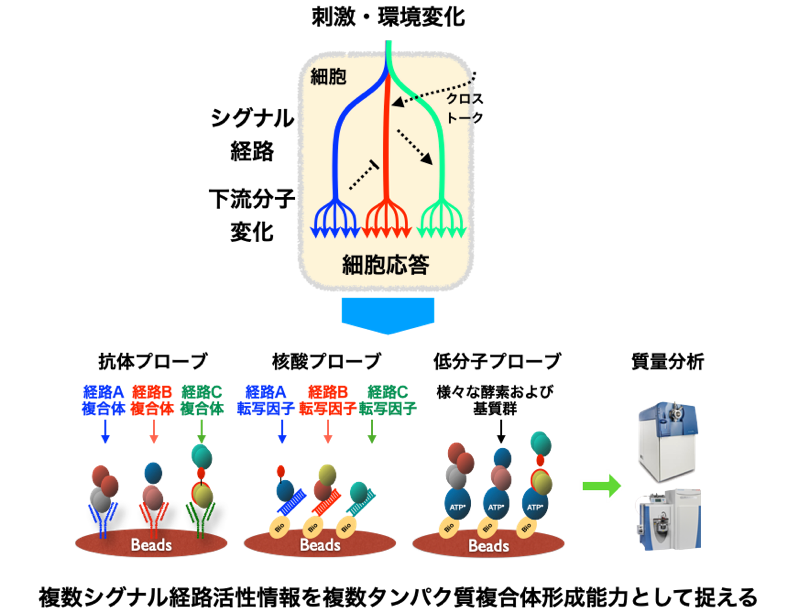 細胞は外界からの刺激や環境変化に対し、ヒトでは300種類以上存在する細胞内シグナル経路の内、複数経路の活性化を介して応答し適応する機構を持っている。細胞応答のメカニズム解明には、刺激に応じて活性化した複数のシグナル経路を明らかにすることが重要であるが、300種類以上のシグナル経路について、個々の経路についての研究は進んでいるものの、網羅的にシグナル経路の活性を測定する手法が存在せず、様々な刺激に対する応答の全体像の把握できず研究のボトルネックとなっている。シグナル経路の活性は、経路に特異的なタンパク質の複合体形成能力で表すことが可能であり、経路特異的なタンパク質に結合する分子プローブ(抗体、核酸、低分子)を用い、複数シグナル経路それぞれに特異的なタンパク質複合体を1tubeで精製し、質量分析により定量することができれば、複数シグナル経路活性の同時計測が可能になると考えられる。我々は、本研究提案でまず10種類のシグナル経路の同時計測技術の開発と検証を行うと共に、その有用性を証明するために 「1種の細胞刺激による複数のシグナル経路の活性化」、「シグナル経路間のクロストーク」に焦点を当て、その意義の解析を行う。
細胞は外界からの刺激や環境変化に対し、ヒトでは300種類以上存在する細胞内シグナル経路の内、複数経路の活性化を介して応答し適応する機構を持っている。細胞応答のメカニズム解明には、刺激に応じて活性化した複数のシグナル経路を明らかにすることが重要であるが、300種類以上のシグナル経路について、個々の経路についての研究は進んでいるものの、網羅的にシグナル経路の活性を測定する手法が存在せず、様々な刺激に対する応答の全体像の把握できず研究のボトルネックとなっている。シグナル経路の活性は、経路に特異的なタンパク質の複合体形成能力で表すことが可能であり、経路特異的なタンパク質に結合する分子プローブ(抗体、核酸、低分子)を用い、複数シグナル経路それぞれに特異的なタンパク質複合体を1tubeで精製し、質量分析により定量することができれば、複数シグナル経路活性の同時計測が可能になると考えられる。我々は、本研究提案でまず10種類のシグナル経路の同時計測技術の開発と検証を行うと共に、その有用性を証明するために 「1種の細胞刺激による複数のシグナル経路の活性化」、「シグナル経路間のクロストーク」に焦点を当て、その意義の解析を行う。
参考文献
- Ninomiya K, Adachi S, Natsume T, Iwakiri J, Terai G, Asai K, Hirose T. LncRNA-dependent nuclear stress bodies promote intron retention through SR protein phosphorylation. EMBO J. Feb 3;39(3):e102729. (2020)
- Takahashi A, Adachi S, Morita M, Tokumasu M, Natsume T, Suzuki T, Yamamoto T. Post-transcriptional Stabilization of Ucp1 mRNA Protects Mice from Diet-Induced Obesity. Cell Rep. Dec 29;13(12):2756-67. (2015)
- Adachi S, Homoto M, Tanaka R, Hioki Y, Murakami H, Suga H, Matsumoto M, Nakayama KI, Hatta T, Iemura S, Natsume T. ZFP36L1 and ZFP36L2 control LDLR mRNA stability via the ERK-RSK pathway. Nucleic Acids Res. Sep;42(15):10037-49. (2014)

